La técnica puede acelerar la identificación de los determinantes de la resistencia de los patógenos resistentes a múltiples fármacos (MDR) y el desarrollo de nuevas estrategias antirresistencia.
La investigación abrió una nueva vía para editar genómicamente esas especies y aislamientos de bacterias silvestres, como los que tienen importancia clínica y medioambiental y los que forman el microbioma humano. También proporcionó un marco para aprovechar otros sistemas CRISPR-Cas generalizados en genomas procarióticos y expandir los kits de herramientas basados en CRISPR. La investigación ha sido publicada en la revista científica líder Investigación de ácidos nucleicos .
El sistema CRISPR-Cas comprende el sistema inmunológico adaptativo en procariotas que desarma a los virus invasores escindiendo su ADN. Debido a su capacidad única de apuntar y alterar secuencias de ADN, CRISPR-Cas se ha aprovechado como el método de edición del genoma de próxima generación.
El método se basa en el sistema CRISPR / Cas9 de clase 2 tipo II, que ha revolucionado la investigación genética y biomédica en una plétora de organismos y fue galardonado con el Premio Nobel de Química 2020. Sin embargo, los sistemas CRISPR-Cas de Clase 2 representan solo ∼10% de los sistemas CRISPR-Cas codificados naturalmente en procariotas. Sus aplicaciones para editar genomas bacterianos son bastante limitadas.
Notablemente, Los sistemas CRISPR-Cas que pertenecen a diferentes clases y tipos se identifican continuamente, y sirven como un depósito profundo para la expansión de los conjuntos de herramientas basados en CRISPR. El sistema CRISPR-Cas más diverso y ampliamente distribuido es el sistema de tipo I que representa el 50% de todos los sistemas CRISPR-Cas identificados y tiene el potencial de expandir los juegos de herramientas basados en CRISPR con ventajas distintivas no accesibles con los sistemas de clase 2. como alta especificidad, desvío mínimo de la orientación, y capaz de deleciones de grandes fragmentos.
Sin embargo, El sistema CRISPR-Cas de tipo I depende de un complejo efector multicomponente denominado Cascade para interferir en el ADN que no es fácilmente transferible a huéspedes heterólogos, obstaculizar la aplicación generalizada de estos CRISPR naturalmente abundantes para la edición del genoma y la terapéutica.
Previamente, el equipo ha identificado un sistema CRISPR-Cas de tipo I-F altamente activo en una clínica resistente a múltiples fármacos P. aeruginosa cepa PA154197 que se aisló de un caso de infección del torrente sanguíneo en el Hospital Queen Mary. Caracterizaron este sistema CRISPR-Cas y desarrollaron con éxito un método de edición del genoma aplicable en el aislado MDR basado en este sistema nativo CRISPR-Cas de tipo I-F. El método permitió la identificación rápida de los determinantes de resistencia del aislado clínico MDR y el desarrollo de una nueva estrategia antirresistencia ( Informes de celda , 2019, 29, 1707-1717).
Para superar la barrera de transferir el complejo tipo I Cascade a huéspedes heterólogos, en este estudio, el equipo clonó todo el tipo I-F cas operón en un vector de integración competente mini-CTX y entregó el casete en huéspedes heterólogos por conjugación, un enfoque de transferencia de ADN común en la naturaleza. El vector mini-CTX permitió la integración de toda la cascada en el conservado attB locus genético en el genoma de los huéspedes heterólogos, permitiéndoles albergar un sistema CRISPR-Cas de tipo I-F "nativo" que puede expresarse y funcionar de manera estable.
El equipo demostró que la cascada de tipo I-F transferida muestra una capacidad de interferencia de ADN significativamente mayor y una mayor estabilidad de cepa que el sistema Cas9 transferible y puede emplearse para la edición del genoma con eficiencia (> 80%) y simplicidad. es decir, mediante una transformación de un solo paso de un solo plásmido de edición.
Es más, desarrollaron un sistema transferible avanzado que incluye tanto una cascada de tipo I-F altamente activa como una recombinasa para promover la aplicación del sistema en cepas con escasa capacidad de recombinación homóloga, salvaje P. aeruginosa aislamientos sin información de la secuencia del genoma, y en otro Pseudomonas especies.
Finalmente, los genes en cascada de tipo I-F introducidos pueden eliminarse fácilmente de los genomas del huésped mediante la eliminación de grandes fragmentos de ADN mediada por la cascada I-F, resultando en la edición del genoma sin cicatrices en las células huésped. También se demostró la aplicación del sistema transferible para la represión de genes, destacando las aplicaciones robustas y diversas del sistema CRISPR transferible tipo I-F desarrollado.
El Dr. Aixin Yan predijo que este nuevo método se extenderá a la edición no solo de patógenos sino también del microbioma para promover la salud humana.
Creemos que la tecnología y las terapias basadas en CRISPR traerán nuevas esperanzas para combatir las superbacterias en el futuro. . "
Dr. Aixin YAN, Profesor adjunto, División de Investigación de Biología Celular y Molecular, Facultad de Ciencias, La Universidad de Hong Kong
 La luz ultravioleta B es buena para el microbioma intestinal
La luz ultravioleta B es buena para el microbioma intestinal
 El perfil bacteriano intestinal puede predecir el daño intestinal después de la radioterapia
El perfil bacteriano intestinal puede predecir el daño intestinal después de la radioterapia
 La terapia biológica puede disminuir el riesgo de COVID-19 grave
La terapia biológica puede disminuir el riesgo de COVID-19 grave
 Infliximab puede obstaculizar la eficacia de algunas vacunas COVID-19
Infliximab puede obstaculizar la eficacia de algunas vacunas COVID-19
 Nueva herramienta registra y rastrea el crecimiento del microbioma
Nueva herramienta registra y rastrea el crecimiento del microbioma
 El nuevo receptor de macrófagos súper activador podría explicar la hiperinflamación en el COVID-19 grave
El nuevo receptor de macrófagos súper activador podría explicar la hiperinflamación en el COVID-19 grave
 Modelo de ratón recién nacido da pistas sobre la causa de una enfermedad intestinal devastadora en bebés prematuros anémicos
Los médicos han sospechado durante mucho tiempo que las transfusiones de glóbulos rojos administradas a bebés prematuros con anemia pueden ponerlos en peligro de desarrollar enterocolitis necrotizante
Modelo de ratón recién nacido da pistas sobre la causa de una enfermedad intestinal devastadora en bebés prematuros anémicos
Los médicos han sospechado durante mucho tiempo que las transfusiones de glóbulos rojos administradas a bebés prematuros con anemia pueden ponerlos en peligro de desarrollar enterocolitis necrotizante
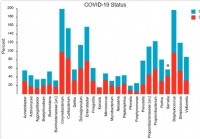 La composición y estructura del microbioma nasofaríngeo se relacionan con la gravedad de la enfermedad COVID-19
Las infecciones virales están asociadas con cambios en el microbioma del tracto respiratorio superior / nasofaríngeo (NP). Además, Muchos estudios plantean la hipótesis de las posibilidades de superin
La composición y estructura del microbioma nasofaríngeo se relacionan con la gravedad de la enfermedad COVID-19
Las infecciones virales están asociadas con cambios en el microbioma del tracto respiratorio superior / nasofaríngeo (NP). Además, Muchos estudios plantean la hipótesis de las posibilidades de superin
 La genética puede influir en la composición del microbioma más que los factores ambientales
Los investigadores que estudian ratones en el Instituto de Tecnología Technion-Israel han descubierto que el microbioma está influenciado por la genética mucho más que por el entorno de nacimiento mat
La genética puede influir en la composición del microbioma más que los factores ambientales
Los investigadores que estudian ratones en el Instituto de Tecnología Technion-Israel han descubierto que el microbioma está influenciado por la genética mucho más que por el entorno de nacimiento mat