Tidligere forskning har afsløret, at CoV'er er tilbøjelige til overførsel af krydsarter. Derfor, indsamlingen af flere oplysninger om dyre -CoV'er er afgørende for at forudsige fremtidige CoV -udbrud og forhindre zoonotiske overførselshændelser.
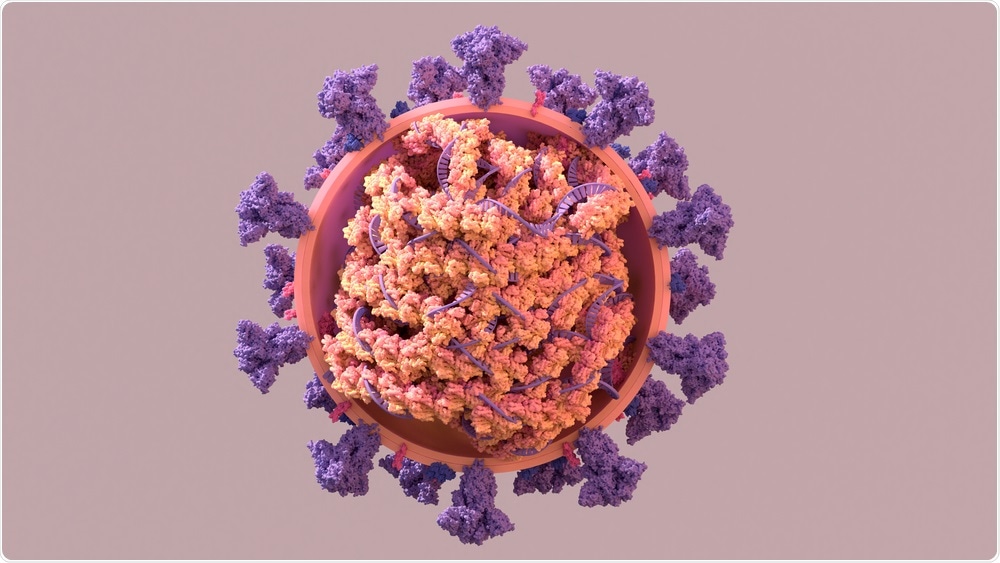 Undersøgelse:En sammenlignende analyse af Coronavirus Nucleocapsid (N) -proteiner afslører SADS-CoV N-proteinet modvirker IFN-β-produktion ved at fremkalde ubiquitination af RIG-I. Billedkredit:Design_Cells / Shutterstock.com
Undersøgelse:En sammenlignende analyse af Coronavirus Nucleocapsid (N) -proteiner afslører SADS-CoV N-proteinet modvirker IFN-β-produktion ved at fremkalde ubiquitination af RIG-I. Billedkredit:Design_Cells / Shutterstock.com
Forskere har for nylig afsløret svin akut diarré syndrom (SADS) -CoV, der tilhører slægten Alphacoronavirus, som et nyt patogen, der forårsager diarré hos nyfødte smågrise. SADS-CoV, som også er kendt som svin enterisk alphacoronavirus (SeACoV), havde rapporteret dødeligheden over 35% i det sydlige Kina under et udbrud i 2017.
Bortset fra SADS-CoV, fire flere svine CoV'er er blevet identificeret til dato; nemlig, overførbar gastroenteritisvirus (TGEV), svin hæmagglutinerende encephalomyelitis virus (PHEV), svinepidemisk diarrévirus (PEDV), og porcine delta coronavirus (PDCoV). Da SADS-CoV er nært beslægtet med bat CoV HKU2-stammer, forskere mener, at denne stamme er opstået som et resultat af genetisk drift eller rekombinationsforekomster mellem co-inficerende CoV'er.
Genomiske undersøgelser har vist, at SADS-CoV omfatter en genetisk sekvens, der består af fire strukturelle proteiner, syv uafhængige åbne læserammer (ORF'er), der koder for seksten ikke-strukturelle proteiner, og et ekstra protein som alle ligner det, der findes i mange CoV'er. Ud af de fire strukturelle proteiner, nucleocapsid (N) -proteinet indeholder en stærkt konserveret genomisk sekvens, der er stærkt udtrykt. N -proteinet spiller en rolle ved virusinfektion og er også involveret i subgenomisk ribonukleinsyre (RNA) transkription, viral genomreplikation, og dets interaktion med andre proteiner for at understøtte virionsamling.
Tidligere undersøgelser har antydet, at SADS-CoV N-proteinet er involveret i virusets unddragelse af værtens medfødte immunrespons, som er kroppens første forsvarslinje mod skadelige patogener. Yderligere, type I interferon (IFN) signalvejen spiller en vigtig rolle i beskyttelsen af værten mod virusinfektion, som inkluderer primær identifikation af patogenassocierede molekylære mønstre (PAMP'er) ved hjælp af mønstergenkendelsesreceptorer (PRR'er).
Ligesom andre RNA -vira, CoV'er producerer PAMP'er, herunder dobbeltstrenget RNA (dsRNA) og 5'-ppp RNA-mellemprodukter i cytoplasmaet under replikation. Disse PAMP'er identificeres derefter ved hjælp af værtsmønstergenkendelsesreceptorer (PRR'er), såsom retinsyre-inducerbare gen I (RIG-I) -lignende receptorer (RLR). Eftergenkendelse og aktivering af RIG-I og/eller melanomdifferentieringsassocieret gen 5 (MDA5) fører til deres interaktion med caspase-aktiverings- og rekrutteringsdomæner (CARD'er).
Efterfølgende, prionlignende polymerer dannes, som stimulerer nedstrøms
TANK -binding kinase 1 (TBK1) og inhibitor af κ B kinase-ϵ (IKKϵ). Aktiveringen af TBK1 fører til phosphorylering af interferon -regulatorisk faktor 3 (IRF3), som, på tur, fremmer produktionen af type I IFN'er. Dette fører i sidste ende til udtryk for hundredvis af IFN-stimulerede gener (ISG'er).
ISG'er udtrykkes på en autokrin og parakrin måde i et forsøg på at beskytte værtscellen mod viral invasion. På trods af disse medfødte forsvar, vira kan ofte udvikle sig for at undgå værtscelleforsvaret. For eksempel, flere CoV'er kan hæmme værts -IFN -responser under infektion.
En nylig undersøgelse offentliggjort i Grænser i immunologi fokuserer på rollerne for SADS-CoV N-proteinet i IFN-undertrykkelse under infektion. I dette studie, forskere sammenlignede aminosyreligheder mellem N -proteiner fra forskellige CoV'er tilhørende fire forskellige slægter. Målene for hvert N -protein, der er forbundet med IFN -signalering, diskuteres også. Mekanismen for IFN-inhibering er blevet bestemt ved hjælp af SADS-CoV N-protein via komparativ analyse.
Forskerne i denne undersøgelse afslørede, at for at undertrykke IFN -signalering, PAMP -genkendelsestrinnet er et kritisk mål for N -proteinet. Til denne ende, interaktionen mellem SADS-CoV N-proteinet og RIG-I udløser ubiquitination, som fremmer proteasomafhængig nedbrydning. Dette fører til undertrykkelse af værtens IFN -svar.
Denne undersøgelse har også evalueret flere N-proteiner af SADS-CoV for at vurdere deres evne til at hæmme IFN-respons. Ultimativt, forskerne fandt ud af, at hæmningen af dette svar ikke er afhængig af aminosyresekvensligheden. For eksempel, der er en 91,2% aminosyrelighed mellem SARS-CoV-2 og SARS-CoV. Imidlertid, hvad angår mekanismerne i spil for SARS-CoV-2, N -proteinet kunne hæmme aktiviteten af IFN -promotoren, som ellers fremkaldes af RIG-I, MAVS, TBK1, og IKKϵ, der henviser til, at SARS-CoV N-protein ikke gjorde det.
Et sådant resultat fremhæver betydningen af tertiær struktur for at definere proteinfunktion. Der er stadig et hul i den nuværende forskning i en komplet forståelse af CoV N -proteinets tertiære struktur. Selvom dette kan være sandt, data om strukturer af N -terminaldomæne (NTD) og C -terminaldomæne (CTD) for forskellige CoV N -proteiner er i øjeblikket tilgængelige.
Resultatet af den aktuelle forskning er på linje med tidligere rapporter. Til denne ende, tidligere undersøgelser har vist, at N -proteinet i PEDV fremkalder værts -IFN -svar ved at interagere med TBK1 direkte. Derudover N-proteinet i SARS-CoV interagerer direkte eller indirekte med TRIM25 og proteinaktivatoren af proteinkinase R (PACT) for at aktivere RIG-I.
Den foreliggende forskning har også antydet, at N-proteinet i SADS-CoV målretter de indledende trin i IFN-responset og kan direkte forstyrre aktiveringen af RIG-I. Denne komparative analyse har også vist, at interferens ved hjælp af RIG-I kan være den førende metode for N-proteinet af SADS-CoV til at undertrykke RIG-I-lignende receptor (RLR) signalering. Denne undersøgelse observerede, at SADS-CoV N-proteinet målretter mod RIG-I for at hæmme IFN-β-promotoraktivitet.
 Tarmbakterier kan forudsige risiko for pulmonal hypertension
Tarmbakterier kan forudsige risiko for pulmonal hypertension
 Fastfood kan være den største synder i teenage -depression
Fastfood kan være den største synder i teenage -depression
 Rotavirus spiller en rolle i udviklingen af type 1 -diabetes
Rotavirus spiller en rolle i udviklingen af type 1 -diabetes
 Nye vært-virale-mikrobiom-interaktioner under COVID-19 kan bestemme resultatet
Nye vært-virale-mikrobiom-interaktioner under COVID-19 kan bestemme resultatet
 Probiotika som adjuvant behandling til COVID-19-patienter
Probiotika som adjuvant behandling til COVID-19-patienter
 Forskning viser, at probiotika kan hjælpe med at bekæmpe angst og depression
Forskning viser, at probiotika kan hjælpe med at bekæmpe angst og depression
 Utæt tarm og mikrobiel dysbiose kan bidrage til cytokinstorm i alvorligt syge COVID-19-tilfælde
Når verden nærmer sig den dystre milepæl med tre millioner dødsfald som følge af COVID-19 sygdom, et nyt fortryk -forskningsartikel, der blev sendt til bioRxiv * server viser, at tilstedeværelsen af
Utæt tarm og mikrobiel dysbiose kan bidrage til cytokinstorm i alvorligt syge COVID-19-tilfælde
Når verden nærmer sig den dystre milepæl med tre millioner dødsfald som følge af COVID-19 sygdom, et nyt fortryk -forskningsartikel, der blev sendt til bioRxiv * server viser, at tilstedeværelsen af
 Plast findes nu almindeligt i afføring hos mennesker
Næsten otte milliarder tons plast finder hvert år vej til havene. Denne enorme mængde plast skyller enten i land eller nedbrydes i små stykker, der er mindre end 5 millimeter i diameter og kaldes mikr
Plast findes nu almindeligt i afføring hos mennesker
Næsten otte milliarder tons plast finder hvert år vej til havene. Denne enorme mængde plast skyller enten i land eller nedbrydes i små stykker, der er mindre end 5 millimeter i diameter og kaldes mikr
 Undersøgelse tyder på en forbindelse mellem probiotisk brug og "hjernetåge"
En undersøgelse foretaget ved Medical College of Georgia ved Augusta University har vist, at probiotisk indtagelse kan resultere i en betydelig ophobning af tyndtarmbakterier, der fører til tåget i hj
Undersøgelse tyder på en forbindelse mellem probiotisk brug og "hjernetåge"
En undersøgelse foretaget ved Medical College of Georgia ved Augusta University har vist, at probiotisk indtagelse kan resultere i en betydelig ophobning af tyndtarmbakterier, der fører til tåget i hj