In silico
analyse en verificatie van de S100 genexpressie bij maagkanker
Abstracte achtergrond
De S100-eiwit familie bestaat uit 22 leden, waarvan eiwitsequenties omvatten ten minste één EF hand Ca
2 + bindend motief. Zij waren betrokken bij de regulatie van verschillende cellulaire processen zoals celcyclus en differentiatie. De uitdrukking status van S100 familieleden bij maagkanker was nog niet bekend.
Methoden
combinatie met analyse van reeksen analyse van genexpressie, virtuele Northern blot en microarray data, de expressieniveaus van S100 familieleden in normale en kwaadaardige maag weefsels werden systematisch onderzocht. De expressie van S100A3 werd verder onderzocht met kwantitatieve RT-PCR.
Resultaten
Minstens 5 S100 genen gevonden in maag cance wordt opgereguleerd door in silico analyse. Onder hen, vier genen, waaronder S100A2, S100A4, S100A7 en S100A10, gemeld dat overexpressie in maagkanker eerder. De expressie van S100A3 in tachtig patiënten van maagkanker werd verder onderzocht. De resultaten toonden aan dat de gemiddelde expressieniveaus van S100A3 bij maagkanker weefsels was 2,5 maal zo hoog als in naburige niet-tumor weefsels. S100A3 expressie werd gecorreleerd met tumor differentiatie en TNM (Tumor-Node-Metastase) stadium van maagkanker, die relatief hoog werd uitgedrukt in slecht gedifferentieerde en gevorderde maagkanker weefsels (P Restaurant < 0,05).
Conclusie
Voor zover wij weten is dit het eerste verslag van een systematische evaluatie van de S100-gen expressies in maagkanker door meerdere in silico analyse. De resultaten gaven aan dat overexpressie van gen-S100 familieleden waren kenmerken van maagkanker en S100A3 zou een belangrijke rol in de differentiatie en de progressie van maagkanker te spelen. Achtergrond
Maagkanker is de tweede meest voorkomende oorzaak van kanker overlijden wereldwijd. Milieu- en genetische factoren zijn beide belangrijk in de maag carcinogenese [1, 2]. In de laatste twee decennia is veel vooruitgang geboekt in het identificeren van genen betrokken bij de ontwikkeling van maagkanker. Deze geïdentificeerde genen zijn ter toelichting van de pathogenese van maagkanker en definiëren de moleculaire signatuur. Ze kunnen ook dienen als biomarkers voor de vroege diagnose en de doelstellingen voor de ontwikkeling van geneesmiddelen.
Onlangs, analyses van grootschalige genexpressie naar voren zijn gekomen als belangrijke instrumenten voor het screenen van genen die in verband met kanker [3]. De twee experimentele technologieën die beschikbaar zijn voor grootschalige genexpressie analyse zijn: 1) DNA-sequencing-gebaseerde seriële analyse van genexpressie (SAGE) en expressed sequence tag (EST) benaderingen en 2) dot-blot gebaseerde microarray-analyse. Meerdere bioinformatica infrastructuur zijn opgericht om gegevens uit deze technieken samen te stellen. Onder hen, de Cancer Genome Project Anatomy (CGAP) en Gene Expression Omnibus (GEO) zijn twee belangrijke netwerken [4, 5]. Eerdere toepassingen datamining gebruik CGAP en GEO middelen hebben geleid tot de identificatie van verschillende nieuwe of bekende kanker-gerelateerde genen [6, 7]. Ondernemingen De S100 eiwitfamilie omvat 22 leden waarvan eiwitsequenties omvatten ten minste een EF hand Ca2 + bindende motief [8]. S100 eiwitten gelokaliseerd in het cytoplasma en /of kern van een groot aantal cellen en betrokken bij de regulatie van een aantal cellulaire processen zoals celcyclus en differentiatie. Zeventien S100 familieleden bevinden zich als een cluster op chromosoom 1q21-22, een regio vaak herschikt in een aantal tumoren. Bovendien heeft moleculaire analyse bleek dat verschillende S100s, waaronder S100A2, S100A4, S100A7 en S100A10, vertonen veranderde expressieniveaus in maagkanker [9, 10]. Het is dus interessant om systematisch onderzoek naar de expressie van andere leden van de S100 familie in normale en maagkanker weefsels.
In deze studie hebben we gebruik gemaakt van de databases en analytische benaderingen verkrijgbaar bij de Gene Expression Omnibus en Cancer Genome Project Anatomy systematisch te analyseren de expressie van 22 genen S100 in normale en kankerachtige weefsels maag. De onafhankelijke SAGE en microarray datasets werden gescreend op S100 genexpressie patronen. We hebben aangetoond dat ten minste vijf S100 genen werden opgereguleerd bij maagkanker en verder experimenteel de opregulatie van S100A3 geverifieerd door kwantitatieve RT-PCR.
Methods
SAGE analyse
SAGE meet het aantal tags die de transcriptie vertegenwoordigen producten van een gen. De door SAGE-technologie data is een lijst van tags met de bijbehorende telling waarden. Alle openbare SAGE gegevens die zijn verzameld in GEO website tot en met januari 2008 werden gebruikt voor de analyse van genexpressie S100. Zowel NlaIII en Sau3A tags uit SAGEmap http:.... //Www NCBI NLM nih gov /SAGE /werden toegewezen aan UniGene clusters http:... //Www NCBI NLM nih . gov /UniGene /. De betrouwbare UniGene clusters afgestemd op S100 labels werden aangenomen. Deze sequentietags werden vervolgens gebruikt om de niveaus van expressie van 22 genen in S100 2 en 8 normale maagkanker bibliotheken bepalen. Een lijst van de labels gebruikt voor analyse werd gegeven in Tabel 1 en gedetailleerde informatie over deze bibliotheken zijn beschikbaar op de website GEO. Analyses werden uitgevoerd door het gemiddelde aantal labels S100 vergelijken in bibliotheken van normale mucosa met die in bibliotheken van maagkanker. Verschil van > 3-voudige verandering zal worden beschouwd als positive.Table 1 SAGE analyse van de S100 genen expressie in normale en maagkanker bibliotheken
Gene naam
Unigene cluster
SAGE tag
Normal (tpm *)
Cancer (tpm*)
S100A2
516484
GATCTCTTGG
0.0
98.1
S100A3
433168
TCTCCCACAC
0.0
2.8
S100A4
81256
ATGTGTAACG
0.0
207.8
S100A6
275243
CCCCCTGGAT
245.9
865.9
S100A7
112408
GAGCAGCGCC
0.0
143.8
S100A8
416073
TACCTGCAGA
0.0
549.2
S100A9
112405
GTGGCCACGG
0.0
637.0
S100A10
143873
AGCAGATCAG
263.6
1890.5
S100A12
19413
GATTTTTAAA
0.0
13.9
S100A16
515714
AGCAGGAGCA
0.0
130.6
Alleen S100s dat de positieve resultaten vertonen, worden in deze tabel weergegeven. * Labels per miljoen;
Virtual Noord
In de CGAP databank, virtuele Northern blotanalyse stelt onderzoekers in staat om de expressie van een specifiek gen te bekijken in alle EST en SAGE bibliotheken [4]. Door middel van de Gene Finder gereedschap http:... //CGAP NCI nih gov /Genes /GeneFinder, wat georganiseerde informatie over een bepaald gen kan worden gevonden door het bevragen van zowel de unieke identifier-gen of een sleutel woord. Inbegrepen in de beschikbare informatie zijn de EST en SAGE vNorthern expressional patronen in alle beschikbare bibliotheken gerangschikt volgens hun weefsel oorsprong. De SAGE verzamelde gegevens CGAP omvatten 5 weefsel bibliotheken en bibliotheken van 2 xenotransplantaat maagkanker, evenals 3 normale bibliotheken van de maag, die gedeeltelijk verschilt van die verzamelde GEO hierboven werden genoemd. S100 genen waarvan de expressie in EST en SAGE bibliotheken van maagkanker was zowel > 3 vouw zoveel die bij gezonde maag werden aangeduid als positieve
Microarray analyse
Momenteel vijf microarray datasets (tabel 2) bevatten. gegevens van normale en maagkanker weefsels waren beschikbaar in GEO website http: //www. NCBI NLM nih gov /projecten /geo /.... De dataset GSE2669, GSE2701 en GSE3438 bijgedragen door Boussioutas A [11], Chen X [12] en Kim S [13] respectievelijk gekozen voor het uitvoeren van microarray-analyse omdat deze drie datasets relatief meer gevallen van normale en maagkanker (10 normale cellen bevatte en 64 kankers voor Boussioutas A et al; 22 normale mensen en 90 kankers voor Chen X; 50 normale mensen en 50 kankers voor Kim S). Meer details over de monsters en microarray analyse kan worden gevonden van de GEO website. Verschil werd significant geacht wanneer p
< 0,05. S100 genen waarvan de expressie werd zowel zeer in drie groepen van maagkanker weefsels werden aangeduid als positief ones.Table 2 microarray datasets van maagkanker opgevangen in Gene Expression Omnibus website.
Cases
Array
Spot
Leverancier
|
Normaal
Cancer
|
|
|
GSE2637
3
55
cDNA
13 k /17 K
Aggarwal A et al
GSE2685
8
22
oligonucleotide
~ 7.2 K
Hippo Y et al
GSE2669
10
|  Wat u eet, kan de manier veranderen waarop antibiotica uw darmen beïnvloeden
Wat u eet, kan de manier veranderen waarop antibiotica uw darmen beïnvloeden
 Op geitenmelk gebaseerde formule goed voor de darmgezondheid van zuigelingen
Op geitenmelk gebaseerde formule goed voor de darmgezondheid van zuigelingen
 Dieet en voeding beïnvloeden het microbioom in het colonslijmvlies
Dieet en voeding beïnvloeden het microbioom in het colonslijmvlies
 Plantaardig voedsel kan antibioticaresistente superbacteriën op mensen overbrengen
Plantaardig voedsel kan antibioticaresistente superbacteriën op mensen overbrengen
 Omgaan met coeliakie
Omgaan met coeliakie
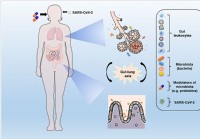 Microbiota-modulatie en herstel van eubiose kunnen helpen om COVID-19-complicaties te beteugelen
Microbiota-modulatie en herstel van eubiose kunnen helpen om COVID-19-complicaties te beteugelen
 Onderzoek toont aan dat darmparasieten de ernst van COVID-19 verminderen
We leren elke dag meer over de ziekte van COVID-19. Volwassenen van elke leeftijd met bepaalde onderliggende medische aandoeningen lopen een verhoogd risico op een ernstige ziekte door het virus dat C
Onderzoek toont aan dat darmparasieten de ernst van COVID-19 verminderen
We leren elke dag meer over de ziekte van COVID-19. Volwassenen van elke leeftijd met bepaalde onderliggende medische aandoeningen lopen een verhoogd risico op een ernstige ziekte door het virus dat C
 Orale seks kan bacteriële vaginose veroorzaken
Een nieuwe studie gepubliceerd in het tijdschrift PLOS Biologie heeft aangetoond dat diverse microbiële populaties vaginale kolonisatie van pathogenen kunnen vergemakkelijken, wat op zijn beurt de k
Orale seks kan bacteriële vaginose veroorzaken
Een nieuwe studie gepubliceerd in het tijdschrift PLOS Biologie heeft aangetoond dat diverse microbiële populaties vaginale kolonisatie van pathogenen kunnen vergemakkelijken, wat op zijn beurt de k
 Voedsel beïnvloedt selectief darmmicroben, vindt onderzoek
Voedsel zou een rol kunnen spelen bij de vorming van de microbiële flora in de menselijke darm. Dit is keer op keer aangetoond in verschillende onderzoeken en onderzoeken. Nu hebben onderzoekers van d
Voedsel beïnvloedt selectief darmmicroben, vindt onderzoek
Voedsel zou een rol kunnen spelen bij de vorming van de microbiële flora in de menselijke darm. Dit is keer op keer aangetoond in verschillende onderzoeken en onderzoeken. Nu hebben onderzoekers van d