"Elle n'avait pratiquement aucun signe avant-coureur ou symptôme, " a dit Poore. " Personne ne pourrait dire pourquoi son cancer n'a pas été détecté plus tôt ou pourquoi il était résistant au traitement qu'ils ont essayé. "
Comme Poore est venu apprendre à travers ses études collégiales, le cancer a traditionnellement été considéré comme une maladie du génome humain -; les mutations de nos gènes permettent aux cellules d'éviter la mort, proliférer et former des tumeurs.
Mais quand Poore a vu une étude de 2017 dans Science qui a montré comment les microbes ont envahi la majorité des cancers du pancréas et ont réussi à décomposer le principal médicament de chimiothérapie administré à ces patients, il était intrigué par l'idée que les bactéries et les virus pourraient jouer un rôle plus important dans le cancer que quiconque ne l'avait envisagé auparavant.
Poore est actuellement un MD/Ph.D. étudiant à la faculté de médecine de l'Université de Californie à San Diego, où il mène ses travaux de thèse dans le laboratoire de Rob Knight, Doctorat., professeur et directeur du Center for Microbiome Innovation.
En collaboration avec un groupe interdisciplinaire de collaborateurs, Poore et Knight ont développé une nouvelle méthode pour identifier qui a le cancer, et souvent de quel type, en analysant simplement les modèles d'ADN microbien - ; bactérienne et virale - ; présent dans leur sang.
L'étude, publié le 11 mars 2020 en La nature , peut changer la façon dont le cancer est perçu, et diagnostiqué.
Presque tous les efforts de recherche sur le cancer antérieurs ont supposé que les tumeurs sont des environnements stériles, et ignoré l'interaction complexe que les cellules cancéreuses humaines peuvent avoir avec les bactéries, virus et autres microbes qui vivent dans et sur notre corps.
Le nombre de gènes microbiens dans notre corps dépasse largement le nombre de gènes humains, il ne devrait donc pas être surprenant qu'ils nous donnent des indices importants sur notre santé."
Rob Chevalier, Doctorat., professeur et directeur du Center for Microbiome Innovation
Les chercheurs ont d'abord examiné les données microbiennes disponibles dans The Cancer Genome Atlas, une base de données de l'Institut national du cancer contenant des informations génomiques et autres de milliers de tumeurs de patients. A la connaissance de l'équipe, il s'agissait du plus grand effort jamais entrepris pour identifier l'ADN microbien dans les données de séquençage humain.
A partir de 18 ans, 116 échantillons de tumeurs, représentant 10, 481 patients avec 33 types de cancer différents, ont émergé des signatures microbiennes distinctes, ou des motifs, associés à des types de cancer spécifiques. Certains étaient attendus, comme l'association entre le papillomavirus humain (HPV) et le col de l'utérus, cancers de la tête et du cou, et l'association entre Fusobactérie espèces et les cancers gastro-intestinaux. Mais l'équipe a également identifié des signatures microbiennes auparavant inconnues qui distinguaient fortement les types de cancer. Par exemple, la présence de Fécalibactérie espèces distinguaient le cancer du côlon des autres cancers.
Armé des profils de microbiome de milliers d'échantillons de cancer, les chercheurs ont ensuite formé et testé des centaines de modèles d'apprentissage automatique pour associer certains modèles microbiens à la présence de cancers spécifiques. Les modèles d'apprentissage automatique ont pu identifier le type de cancer d'un patient en utilisant uniquement les données microbiennes de son sang.
Les chercheurs ont ensuite retiré les cancers de haut grade (stades III et IV) de l'ensemble de données et ont découvert que de nombreux types de cancer étaient encore distinguables à des stades antérieurs en se fondant uniquement sur des données microbiennes dérivées du sang. Les résultats se sont maintenus même lorsque l'équipe a effectué la décontamination bioinformatique la plus stricte sur les échantillons, qui a supprimé plus de 90 pour cent des données microbiennes.
Pour déterminer si ces modèles microbiens pourraient être utiles dans le monde réel, Chevalier, Poore et son équipe ont analysé des échantillons de plasma dérivés du sang de 59 patients consentants atteints d'un cancer de la prostate, 25 avec un cancer du poumon et 16 avec un mélanome, fournis par des collaborateurs du Moores Cancer Center de l'UC San Diego Health. En utilisant de nouveaux outils qu'ils ont développés pour minimiser la contamination, les chercheurs ont développé une lecture des signatures microbiennes pour chaque échantillon de patient cancéreux et les ont comparées entre elles et avec des échantillons de plasma de 69 patients sains, volontaires séronégatifs, fourni par le HIV Neurobehavioral Research Center de l'UC San Diego School of Medicine.
Les modèles d'apprentissage automatique de l'équipe ont permis de distinguer la plupart des personnes atteintes de cancer de celles qui n'en ont pas. Par exemple, les modèles pourraient identifier correctement une personne atteinte d'un cancer du poumon avec une sensibilité de 86 pour cent et une personne sans maladie pulmonaire avec une spécificité de 100 pour cent. Ils pouvaient souvent dire quels participants avaient lequel des trois types de cancer. Par exemple, les modèles pouvaient correctement distinguer une personne atteinte d'un cancer de la prostate d'une personne atteinte d'un cancer du poumon avec une sensibilité de 81 %.
"La capacité, dans un seul tube de sang, avoir un profil complet de l'ADN de la tumeur (nature) ainsi que de l'ADN du microbiote du patient (nurture), pour ainsi dire, est un pas en avant important pour mieux comprendre les interactions hôte-environnement dans le cancer, " a déclaré le co-auteur Sandip Pravin Patel, MARYLAND, un oncologue médical et co-responsable des thérapies expérimentales au Moores Cancer Center de l'UC San Diego Health.
« Avec cette approche, il est possible de surveiller ces changements au fil du temps, non seulement comme diagnostic, mais pour un suivi thérapeutique à long terme. Cela pourrait avoir des implications majeures pour la prise en charge des patients atteints de cancer, et dans la détection précoce du cancer, si ces résultats continuent à tenir dans d'autres tests."
Selon Patel, le diagnostic de la plupart des cancers nécessite actuellement une biopsie chirurgicale ou le prélèvement d'un échantillon du site suspecté du cancer et l'analyse de l'échantillon par des experts qui recherchent des marqueurs moléculaires associés à certains cancers. Cette approche peut être invasive, chronophage et coûteux.
Plusieurs sociétés développent actuellement des « biopsies liquides » ; des méthodes pour diagnostiquer rapidement des cancers spécifiques à l'aide d'une simple prise de sang et des technologies qui leur permettent de détecter des mutations génétiques humaines spécifiques au cancer dans l'ADN circulant libéré par les tumeurs. Cette approche peut déjà être utilisée pour suivre la progression des tumeurs pour certains types de cancers déjà diagnostiqués, mais n'est pas encore approuvé par la Food and Drug Administration (FDA) des États-Unis pour un usage diagnostique.
« Bien qu'il y ait eu des progrès incroyables dans le domaine de la biopsie liquide et de la détection précoce du cancer, les biopsies liquides actuelles ne sont pas encore capables de distinguer de manière fiable la variation génétique normale du véritable cancer précoce, et ils ne peuvent pas détecter les cancers où les altérations génomiques humaines ne sont pas connues ou ne sont pas détectables, " dit Patel, qui est également directeur adjoint du San Diego Center for Precision Immunotherapy.
C'est pourquoi il existe souvent un risque que les biopsies liquides actuelles donnent des résultats faussement négatifs dans un contexte de faible charge de morbidité. "Il est difficile de trouver une mutation génétique humaine très rare dans une cellule rare provenant d'une tumeur, " A déclaré Patel. "Ils sont faciles à ignorer et on pourrait vous dire que vous n'avez pas de cancer, quand tu le fais vraiment."
Selon les chercheurs, un avantage de la détection du cancer basée sur l'ADN microbien, par rapport à l'ADN tumoral humain circulant, est sa diversité entre les différents sites du corps. ADN humain, en revanche, est essentiellement le même dans tout le corps. En ne s'appuyant pas sur de rares modifications de l'ADN humain, l'étude suggère que les lectures d'ADN microbien à base de sang peuvent être en mesure de détecter avec précision la présence et le type de cancers à des stades plus précoces que les tests actuels de biopsie liquide, ainsi que pour les cancers dépourvus de mutations génétiques détectables par ces plateformes.
Les chercheurs n'hésitent pas à souligner qu'il existe toujours la possibilité que les lectures d'ADN microbien à base de sang puissent manquer des signes de cancer et renvoyer un résultat faussement négatif. Mais ils s'attendent à ce que leur nouvelle approche devienne plus précise à mesure qu'ils affinent leurs modèles d'apprentissage automatique avec plus de données.
Et tandis que les faux négatifs peuvent être moins fréquents avec l'approche de l'ADN microbien, faux positifs -; entendre que vous avez un cancer alors que vous n'en avez pas - ; sont toujours un risque.
Patel a dit que juste parce qu'un cancer est détecté tôt, cela ne signifie pas qu'il nécessite toujours un traitement immédiat. Certaines modifications de l'ADN ne sont pas cancéreuses, changements liés au vieillissement, inoffensif ou auto-résolu. Vous ne les sauriez jamais sans le test. C'est pourquoi plus de dépistage et plus de diagnostics de cancer ne sont pas toujours une bonne chose, Patel a dit, et doit être déterminé par des cliniciens experts.
L'équipe a également averti que même si une lecture microbienne indique un cancer, le patient aurait probablement besoin de tests supplémentaires pour confirmer le diagnostic, déterminer le stade de la tumeur et identifier son emplacement exact.
Knight a déclaré que de nombreux défis restaient à relever alors que son équipe développe davantage ces observations initiales en un test de diagnostic du cancer approuvé par la FDA. Surtout, ils doivent valider leurs résultats auprès d'une population de patients beaucoup plus vaste et diversifiée, une entreprise coûteuse. Ils doivent définir à quoi pourrait ressembler une lecture microbienne "saine" à base de sang parmi beaucoup d'autres, personnes diverses. Ils aimeraient également déterminer si les signatures microbiennes qu'ils peuvent détecter dans le sang humain proviennent de microbes vivants, des microbes morts ou des microbes morts qui ont éclaté, disperser leur contenu - ; un aperçu qui pourrait les aider à affiner et à améliorer leur approche.
Pour faire progresser les lectures d'ADN microbien dans le sang à travers les prochaines étapes vers l'approbation réglementaire, commercialisation et application clinique d'un test de diagnostic, Knight et Poore ont déposé des demandes de brevet et ils ont fondé une entreprise dérivée appelée Micronoma, avec la co-auteur Sandrine Miller-Montgomery, Doctorat, professeur de pratique à la Jacobs School of Engineering et directeur exécutif du Center for Microbiome Innovation de l'UC San Diego.
La dernière étude peut provoquer des changements importants dans le domaine de la biologie du cancer, dit Poore.
"Par exemple, il est courant pour les microbiologistes d'utiliser de nombreux contrôles de contamination dans leurs expériences, mais ceux-ci ont été historiquement rarement utilisés dans les études sur le cancer, " a-t-il dit. " Nous espérons que cette étude encouragera les futurs chercheurs sur le cancer à être " conscients des microbes ". "
Les chercheurs suggèrent également que le diagnostic du cancer n'est peut-être que le début du microbiome sanguin associé au cancer récemment découvert.
« Cette nouvelle compréhension de la façon dont les populations microbiennes évoluent avec le cancer pourrait ouvrir une toute nouvelle voie thérapeutique, " a déclaré Miller-Montgomery. " Nous savons maintenant que les microbes sont là, mais que font-ils ? Et pourrions-nous manipuler ou imiter ces microbes pour traiter le cancer ?"
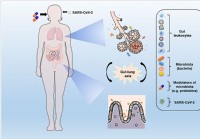 La modulation du microbiote et le rétablissement de l'eubiose pourraient aider à freiner les complications du COVID-19
La modulation du microbiote et le rétablissement de l'eubiose pourraient aider à freiner les complications du COVID-19
 La greffe de liquide vaginal pourrait aider à traiter la vaginose bactérienne récurrente
La greffe de liquide vaginal pourrait aider à traiter la vaginose bactérienne récurrente
 Le microbiome intestinal est également une réalité dans la vie fœtale
Le microbiome intestinal est également une réalité dans la vie fœtale
 Le microbiome intestinal pourrait jouer un rôle dans le COVID-19 sévère
Le microbiome intestinal pourrait jouer un rôle dans le COVID-19 sévère
 Vas-y,
Vas-y,
 Selon une étude, les aliments affectent sélectivement les microbes intestinaux
Selon une étude, les aliments affectent sélectivement les microbes intestinaux
 Le bain de bouche a un impact sur les effets de l'exercice
Les scientifiques savent depuis longtemps que la pression artérielle chute après lexercice, mais le mécanisme na pas été entièrement élucidé. Une nouvelle étude montre que les bactéries dans la bouche
Le bain de bouche a un impact sur les effets de l'exercice
Les scientifiques savent depuis longtemps que la pression artérielle chute après lexercice, mais le mécanisme na pas été entièrement élucidé. Une nouvelle étude montre que les bactéries dans la bouche
 Pourquoi devriez-vous inclure des sources naturelles de fibres dans votre alimentation
Lintérêt pour les régimes à faible teneur en glucides tels que le régime cétogène ou Atkins sest accru ces dernières années. Cependant, comme les mauvaises graisses et les bonnes graisses, les glucide
Pourquoi devriez-vous inclure des sources naturelles de fibres dans votre alimentation
Lintérêt pour les régimes à faible teneur en glucides tels que le régime cétogène ou Atkins sest accru ces dernières années. Cependant, comme les mauvaises graisses et les bonnes graisses, les glucide
 Comprendre les brûlures d'estomac
Brûlures destomac, la sensation de brûlure qui se produit soit dans la poitrine, soit dans la gorge, est généralement un problème temporaire - peut-être avez-vous mangé de la nourriture épicée - mais
Comprendre les brûlures d'estomac
Brûlures destomac, la sensation de brûlure qui se produit soit dans la poitrine, soit dans la gorge, est généralement un problème temporaire - peut-être avez-vous mangé de la nourriture épicée - mais