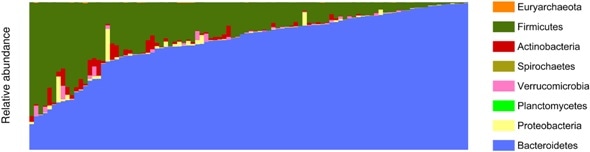
 Il grafico a barre impilato della composizione filogenetica di tutti i taxa del microbioma in questo studio è crollato a livello di phyla nei campioni fecali. Le barre verdi rappresentano Firmicutes e il blu rappresenta Bacteroidetes, le due famiglie batteriche più abbondanti. Ai fini estetici i campioni (n =98, in basso) sono stati ordinati in base alla loro composizione di Bacteroidetes e Firmicutes per dimostrare come i risultati del microbioma intestinale di base di questo studio potrebbero essere utilizzati insieme ai risultati di studi precedenti. Credito:King et al, 2019
Il grafico a barre impilato della composizione filogenetica di tutti i taxa del microbioma in questo studio è crollato a livello di phyla nei campioni fecali. Le barre verdi rappresentano Firmicutes e il blu rappresenta Bacteroidetes, le due famiglie batteriche più abbondanti. Ai fini estetici i campioni (n =98, in basso) sono stati ordinati in base alla loro composizione di Bacteroidetes e Firmicutes per dimostrare come i risultati del microbioma intestinale di base di questo studio potrebbero essere utilizzati insieme ai risultati di studi precedenti. Credito:King et al, 2019 Sebbene l'interesse della ricerca sull'importanza del microbioma intestinale umano per la salute generale continui a crescere, attualmente non esiste un elenco completo di riferimento del microbioma intestinale disponibile per ricercatori e pazienti. In questo studio, King e colleghi iniziano a catalogare la composizione organica dei microbiomi intestinali umani sani, e ha sviluppato un prototipo di modello di refertazione per consentire ai medici di trasmettere i risultati ai pazienti.
Per compilare il loro database, gli autori hanno sequenziato geneticamente 48 campioni fecali di sedici partecipanti sani reclutati dal campus della George Washington University a Washington, DC, oltre a utilizzare 50 campioni metagenomici fecali scaricati dal Progetto Microbioma Umano da individui selezionati come "sani".
Dopo aver analizzato le sequenze genomiche e metagenomiche di tutti i campioni utilizzando un nuovo flusso di lavoro basato su software, King e colleghi hanno compilato un database iniziale di microbi confermati e la loro relativa abbondanza in tutti i campioni, il GutFeelingKB, utilizzando le informazioni genetiche complete e pubblicamente disponibili dell'NCBI per fornire metadati sugli organismi descritti.
Il GutFeelingKB descrive 157 organismi (155 batteri e due organismi archeali) in 60 generi distinti. Il più grande phylum di batteri rappresentato era Firmicutes (40 percento di tutti gli organismi nell'elenco), che a sua volta era costituito per il 20 per cento da Clostridi, 19 percento batterioidi, 17% Bifidobatteri, 14% di enterobatteri, e il 14% di batteri Lactobacillales, classi di batteri presenti anche nello yogurt e in altri alimenti probiotici. Gli autori notano anche che 84 organismi erano comuni a tutti i campioni, potenzialmente indicando che queste potrebbero essere specie chiave per l'intestino umano.
Questo studio ha reclutato solo sedici partecipanti, un piccolo campione. Ma mentre ulteriori studi potrebbero continuare a identificare ulteriori organismi presenti in intestini sani da tutto il mondo, GutFeelingKB è un primo passo importante. Il database potrebbe fungere da punto di partenza per l'analisi comparativa dei campioni e lo sviluppo di futuri trattamenti con il microbioma dei pazienti.
Gli autori aggiungono:
Il nostro obiettivo è mappare il microbioma intestinale sano in modo che noi e altri ricercatori possiamo utilizzare i nostri dati per sviluppare modelli di previsione specifici della malattia".
 Il pet food crudo è un rischio per l'uomo e gli animali
Il pet food crudo è un rischio per l'uomo e gli animali
 Il microbioma umano elimina i glicani della mucosa,
Il microbioma umano elimina i glicani della mucosa,
 I microbiomi intestinali e orali predicono la gravità del COVID-19
I microbiomi intestinali e orali predicono la gravità del COVID-19
 Il modello di topo appena nato fornisce indizi sulla causa di una devastante malattia intestinale nei prematuri anemici
Il modello di topo appena nato fornisce indizi sulla causa di una devastante malattia intestinale nei prematuri anemici
 Gli scienziati risolvono il misterioso caso di sindrome da birrificio automobilistico
Gli scienziati risolvono il misterioso caso di sindrome da birrificio automobilistico
 Le cellule immunitarie intestinali potrebbero essere responsabili dei cambiamenti del metabolismo secondo uno studio
Le cellule immunitarie intestinali potrebbero essere responsabili dei cambiamenti del metabolismo secondo uno studio
 Il cibo colpisce selettivamente i microbi intestinali trova studio
Il cibo potrebbe svolgere un ruolo nella formazione della flora microbica allinterno dellintestino umano. Questo è stato dimostrato più volte in diversi studi e ricerche. Ora i ricercatori della San D
Il cibo colpisce selettivamente i microbi intestinali trova studio
Il cibo potrebbe svolgere un ruolo nella formazione della flora microbica allinterno dellintestino umano. Questo è stato dimostrato più volte in diversi studi e ricerche. Ora i ricercatori della San D
 I probiotici possono aiutare a frenare la malnutrizione nei prossimi due decenni,
dice Bill Gates I probiotici o i batteri buoni hanno mostrato risultati promettenti nel mantenere un intestino sano. Molti studi hanno fornito prove sui benefici per la salute dei probiotici. Ora, il
I probiotici possono aiutare a frenare la malnutrizione nei prossimi due decenni,
dice Bill Gates I probiotici o i batteri buoni hanno mostrato risultati promettenti nel mantenere un intestino sano. Molti studi hanno fornito prove sui benefici per la salute dei probiotici. Ora, il
 Le restrizioni COVID-19 hanno portato a un calo dell'86 percento delle infezioni da norovirus negli Stati Uniti,
trova studio Linizio della pandemia di COVID-19 ha portato allimposizione di numerosi interventi non farmaceutici (NPI) in tutto il mondo. Mentre i cittadini negli Stati Uniti si sono irritati per il
Le restrizioni COVID-19 hanno portato a un calo dell'86 percento delle infezioni da norovirus negli Stati Uniti,
trova studio Linizio della pandemia di COVID-19 ha portato allimposizione di numerosi interventi non farmaceutici (NPI) in tutto il mondo. Mentre i cittadini negli Stati Uniti si sono irritati per il