O novo método identifica grupos de bactérias relevantes do ponto de vista médico e ecológico.
Identificar espécies entre plantas e animais tem sido uma ocupação de tempo integral para alguns biólogos, mas a tarefa é ainda mais difícil para a miríade de micróbios que habitam o planeta. Agora, Os pesquisadores do MIT desenvolveram uma medição simples do fluxo gênico que pode definir populações ecologicamente importantes entre bactérias e arquéias, incluindo identificar populações associadas a doenças humanas.
A métrica de fluxo gênico separa micróbios coexistentes em populações geneticamente e ecologicamente distintas, Martin Polz, professor de engenharia civil e ambiental no MIT, e colegas escrevem na edição de 8 de agosto da
Célula. Polz e seus colegas também desenvolveram um método para identificar partes do genoma nessas populações que apresentam diferentes adaptações que podem ser mapeadas em diferentes ambientes. Quando eles testaram sua abordagem em uma bactéria intestinal, por exemplo, eles foram capazes de determinar que diferentes populações da bactéria estavam associadas a indivíduos saudáveis e pacientes com doença de Crohn.
Os biólogos costumam chamar um grupo de plantas ou animais de espécie se o grupo estiver reprodutivamente isolado de outros - isto é, os indivíduos do grupo podem se reproduzir uns com os outros, mas eles não podem se reproduzir com outros. Como resultado, membros de uma espécie compartilham um conjunto de genes que difere de outras espécies. Grande parte da teoria da evolução centra-se em espécies e populações, os representantes de uma espécie em uma determinada área.
Mas os micróbios "desafiam o conceito clássico de espécie para plantas e animais, "Polz explica. Micróbios tendem a se reproduzir assexuadamente, simplesmente se dividindo em dois, em vez de combinar seus genes com outros indivíduos para produzir descendentes. Os micróbios também são notórios por "pegar DNA de fontes ambientais, como vírus, "ele diz." Os vírus podem transferir DNA para células microbianas e esse DNA pode ser incorporado em seus genomas.
Esses processos tornam difícil classificar os micróbios coexistentes em populações distintas com base em sua composição genética. "Se não podemos identificar essas populações de micróbios, não podemos aplicar, um a um, toda esta rica teoria ecológica e evolutiva que foi desenvolvida para plantas e animais aos micróbios, "diz Polz.
Se os pesquisadores quiserem medir a resiliência de um ecossistema em face das mudanças ambientais, por exemplo, eles podem observar como as populações dentro das espécies mudam ao longo do tempo. "Se não sabemos o que é uma espécie, é muito difícil medir e avaliar esses tipos de perturbações, " ele adiciona.
Um parâmetro para o fluxo gênico Martin e seus colegas decidiram procurar outra maneira de definir populações ecologicamente significativas em micróbios. Liderado pelo estudante de graduação em microbiologia Philip Arevalo, os pesquisadores desenvolveram uma métrica de fluxo gênico que eles chamaram de PopCOGenT (Populations as Clusters Of Gene Transfer).
O PopCOGenT mede o fluxo gênico recente ou a transferência gênica entre genomas intimamente relacionados. Em geral, Os genomas microbianos que trocaram DNA recentemente devem compartilhar trechos mais longos e mais frequentes de DNA idêntico do que se os indivíduos estivessem apenas se reproduzindo ao dividir seu DNA em dois. Sem esse tipo de troca recente, os pesquisadores sugeriram, o comprimento desses trechos compartilhados de DNA idêntico diminuiria à medida que as mutações inserem novas "letras" no trecho.
Duas cepas microbianas que não são geneticamente idênticas uma à outra, mas compartilham "pedaços" consideráveis de DNA idêntico, provavelmente estão trocando mais material genético entre si do que com outras cepas. Esta medição de fluxo gênico pode definir populações microbianas distintas, como os pesquisadores descobriram em seus testes de três tipos diferentes de bactérias.
No
Vibrio bactérias, por exemplo, populações intimamente relacionadas podem compartilhar algumas sequências de genes centrais, mas eles parecem completamente isolados uns dos outros quando vistos através desta medição do fluxo gênico recente, Polz e seus colegas descobriram.
Polz diz que o método PopCOGenT pode funcionar melhor na definição de populações microbianas do que estudos anteriores porque se concentra no fluxo gênico recente entre organismos intimamente relacionados, em vez de incluir eventos de fluxo gênico que podem ter acontecido há milhares de anos.
O método também sugere que, embora os micróbios estejam constantemente absorvendo DNA diferente de seu ambiente que pode obscurecer os padrões de fluxo gênico, "pode ser que esse DNA divergente seja realmente removido pela seleção de populações muito rapidamente, "diz Polz.
A abordagem da ecologia reversa O aluno de pós-graduação em microbiologia David VanInsberghe sugeriu então uma abordagem de "ecologia reversa" que poderia identificar regiões do genoma nessas populações recém-definidas que mostram "varreduras seletivas" - lugares onde a variação do DNA é reduzida ou eliminada, provavelmente como resultado de uma forte seleção natural para uma determinada variante genética benéfica.
Ao identificar varreduras específicas dentro das populações, e mapeando a distribuição dessas populações, o método pode revelar possíveis adaptações que levam os micróbios a habitar um determinado ambiente ou hospedeiro - sem qualquer conhecimento prévio de seu ambiente. Quando os pesquisadores testaram essa abordagem na bactéria intestinal
Ruminococcus gnavus , eles descobriram populações separadas do micróbio associado a pessoas saudáveis e pacientes com doença de Crohn.
Polz diz que o método de ecologia reversa provavelmente será aplicado em um futuro próximo para estudar toda a diversidade das bactérias que habitam o corpo humano. "Há muito interesse no sequenciamento de organismos intimamente relacionados dentro do microbioma humano e na busca por associações de saúde e doenças, e os conjuntos de dados estão crescendo. "
Ele espera usar a abordagem para examinar o "genoma flexível" dos micróbios. Cepas de
E. coli bactérias, por exemplo, compartilham cerca de 40 por cento de seus genes em um "genoma central, "enquanto os outros 60 por cento - a parte flexível - variam entre as cepas." Para mim, é uma das maiores questões da microbiologia:por que esses genomas são tão diversos em conteúdo genético? ", explica Polz." Uma vez que podemos definir as populações como unidades evolutivas, podemos interpretar as frequências gênicas nessas populações à luz dos processos evolutivos. "
As descobertas de Polz e colegas podem aumentar as estimativas da diversidade de micróbios, diz Marx. "O que eu acho muito legal sobre essa abordagem do grupo de Martin é que eles realmente sugerem que a complexidade que vemos é ainda mais complexa do que acreditamos. Pode haver ainda mais tipos que são ecologicamente importantes por aí, coisas que, se fossem plantas e animais, estaríamos chamando-os de espécies. "
Outros autores do MIT no papel incluem Joseph Elsherbini e Jeff Gore. A pesquisa foi apoiada, em parte, pela National Science Foundation e pela Simons Foundation.
 Bactérias no canal do parto associadas à redução do risco de câncer de ovário
Bactérias no canal do parto associadas à redução do risco de câncer de ovário
 Probióticos como terapia adjuvante para pacientes COVID-19
Probióticos como terapia adjuvante para pacientes COVID-19
 A pesquisa mostra como os micróbios intestinais afetam a gripe estomacal
A pesquisa mostra como os micróbios intestinais afetam a gripe estomacal
 A genética pode influenciar a composição do microbioma mais do que fatores ambientais
A genética pode influenciar a composição do microbioma mais do que fatores ambientais
 Abrindo um Esôfago Estreito
Abrindo um Esôfago Estreito
 Bactéria intestinal associada a alterações metabólicas e autismo em novo estudo
Bactéria intestinal associada a alterações metabólicas e autismo em novo estudo
 Esse Pepto provavelmente não ajudará sua úlcera
A úlcera péptica é uma ferida ou lesão que se desenvolve no revestimento do esôfago, estômago, ou duodeno. Não faz muito tempo, presumia-se que as úlceras eram causadas por uma pessoa estressada ou si
Esse Pepto provavelmente não ajudará sua úlcera
A úlcera péptica é uma ferida ou lesão que se desenvolve no revestimento do esôfago, estômago, ou duodeno. Não faz muito tempo, presumia-se que as úlceras eram causadas por uma pessoa estressada ou si
 Bactéria intestinal ligada a músculos mais fortes em pessoas mais velhas
Um novo estudo ousado sugere que a força muscular em adultos mais velhos pode ser maior devido a, em parte, mecanismos envolvendo bactérias intestinais. p As bactérias intestinais estão relacionadas
Bactéria intestinal ligada a músculos mais fortes em pessoas mais velhas
Um novo estudo ousado sugere que a força muscular em adultos mais velhos pode ser maior devido a, em parte, mecanismos envolvendo bactérias intestinais. p As bactérias intestinais estão relacionadas
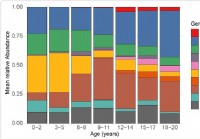 Alteração do microbioma respiratório superior em crianças relacionada à suscetibilidade à SARS-CoV-2
Os pesquisadores testaram o microbioma nasofaríngeo de crianças de até 21 anos e descobriram que o microbioma muda com a idade. Bactérias específicas, cuja abundância também muda com a idade, estão as
Alteração do microbioma respiratório superior em crianças relacionada à suscetibilidade à SARS-CoV-2
Os pesquisadores testaram o microbioma nasofaríngeo de crianças de até 21 anos e descobriram que o microbioma muda com a idade. Bactérias específicas, cuja abundância também muda com a idade, estão as