Un nuovo metodo identifica gruppi di batteri rilevanti dal punto di vista ecologico e medico.
L'identificazione delle specie tra piante e animali è stata un'occupazione a tempo pieno per alcuni biologi, ma il compito è ancora più arduo per la miriade di microbi che abitano il pianeta. Ora, I ricercatori del MIT hanno sviluppato una semplice misurazione del flusso genico che può definire popolazioni ecologicamente importanti tra batteri e archaea, compresa l'individuazione di popolazioni associate a malattie umane.
La metrica del flusso genico separa i microbi coesistenti in popolazioni geneticamente ed ecologicamente distinte, Martin Polz, un professore di ingegneria civile e ambientale al MIT, e colleghi scrivono nel numero dell'8 agosto di
Cellula. Polz ei suoi colleghi hanno anche sviluppato un metodo per identificare parti del genoma in queste popolazioni che mostrano adattamenti diversi che possono essere mappati su ambienti diversi. Quando hanno testato il loro approccio su un batterio intestinale, ad esempio, sono stati in grado di determinare che diverse popolazioni di batteri erano associate a individui sani e pazienti con malattia di Crohn.
I biologi spesso chiamano un gruppo di piante o animali una specie se il gruppo è isolato dal punto di vista riproduttivo dagli altri, cioè gli individui del gruppo possono riprodursi tra loro, ma non possono riprodursi con altri. Di conseguenza, i membri di una specie condividono un insieme di geni che differiscono dalle altre specie. Gran parte della teoria evoluzionistica è incentrata su specie e popolazioni, i rappresentanti di una specie in una determinata area.
Ma i microbi "sfidano il classico concetto di specie per piante e animali, " spiega Polz. I microbi tendono a riprodursi asessualmente, semplicemente dividendosi in due piuttosto che combinare i loro geni con altri individui per produrre prole. I microbi sono anche noti per "prendere il DNA da fonti ambientali, come virus, " dice. "I virus possono trasferire il DNA nelle cellule microbiche e quel DNA può essere incorporato nei loro genomi".
Questi processi rendono difficile classificare i microbi coesistenti in popolazioni distinte in base al loro corredo genetico. "Se non riusciamo a identificare quelle popolazioni nei microbi, non possiamo applicare direttamente ai microbi tutta questa ricca teoria ecologica ed evolutiva che è stata sviluppata per piante e animali, "dice Polz.
Se i ricercatori vogliono misurare la resilienza di un ecosistema di fronte al cambiamento ambientale, ad esempio, potrebbero osservare come le popolazioni all'interno delle specie cambiano nel tempo. "Se non sappiamo cos'è una specie, è molto difficile misurare e valutare questo tipo di perturbazioni, " Aggiunge.
Un metro per il flusso genico Martin ei suoi colleghi hanno deciso di cercare un altro modo per definire popolazioni ecologicamente significative nei microbi. Guidato dallo studente laureato in microbiologia Philip Arevalo, i ricercatori hanno sviluppato una metrica del flusso genico che hanno chiamato PopCOGenT (Popolazioni come cluster di trasferimento genico).
PopCOGenT misura il flusso genico recente o il trasferimento genico tra genomi strettamente correlati. Generalmente, I genomi microbici che hanno scambiato il DNA di recente dovrebbero condividere tratti più lunghi e più frequenti di DNA identico che se gli individui si stessero riproducendo semplicemente dividendo il loro DNA in due. Senza questo tipo di scambio recente, i ricercatori hanno suggerito, la lunghezza di questi tratti condivisi di DNA identico si accorcia man mano che le mutazioni inseriscono nuove "lettere" nel tratto.
Due ceppi microbici che non sono geneticamente identici tra loro ma condividono "pezzi" considerevoli di DNA identico stanno probabilmente scambiando più materiale genetico tra loro che con altri ceppi. Questa misurazione del flusso genico può definire popolazioni microbiche distinte, come hanno scoperto i ricercatori nei loro test su tre diversi tipi di batteri.
In
Vibrio batteri, ad esempio, popolazioni strettamente imparentate possono condividere alcune sequenze geniche fondamentali, ma sembrano completamente isolati l'uno dall'altro se visti attraverso questa misurazione del flusso genico recente, Polz e colleghi hanno trovato.
Polz afferma che il metodo PopCOGenT può funzionare meglio nella definizione delle popolazioni microbiche rispetto agli studi precedenti perché si concentra sul recente flusso genico tra organismi strettamente correlati, piuttosto che includere eventi di flusso genico che potrebbero essere accaduti migliaia di anni nel passato.
Il metodo suggerisce anche che mentre i microbi assorbono costantemente DNA diverso dal loro ambiente che potrebbe oscurare i modelli di flusso genico, "può darsi che questo DNA divergente venga davvero rimosso dalla selezione dalle popolazioni molto rapidamente, "dice Polz.
L'approccio dell'ecologia inversa Lo studente laureato in microbiologia David VanInsberghe ha quindi suggerito un approccio di "ecologia inversa" che potrebbe identificare le regioni del genoma in queste popolazioni appena definite che mostrano "spostamenti selettivi" - luoghi in cui la variazione del DNA viene ridotta o eliminata, probabilmente a causa di una forte selezione naturale per una particolare variante genetica benefica.
Identificando spazzamenti specifici all'interno delle popolazioni, e mappare la distribuzione di queste popolazioni, il metodo può rivelare possibili adattamenti che spingono i microbi ad abitare un particolare ambiente o ospite, senza alcuna conoscenza preliminare del loro ambiente. Quando i ricercatori hanno testato questo approccio nel batterio intestinale
Ruminococcus gnavus , hanno scoperto popolazioni separate del microbo associate a persone sane e pazienti con malattia di Crohn.
Polz afferma che il metodo dell'ecologia inversa verrà probabilmente applicato nel prossimo futuro per studiare l'intera diversità dei batteri che popolano il corpo umano. "C'è molto interesse nel sequenziare organismi strettamente correlati all'interno del microbioma umano e nella ricerca di associazioni di salute e malattia, e i set di dati stanno crescendo."
Spera di utilizzare l'approccio per esaminare il "genoma flessibile" dei microbi. ceppi di
E. coli batteri, ad esempio, condividono circa il 40% dei loro geni in un "genoma centrale, " mentre l'altro 60 percento - la parte flessibile - varia tra i ceppi. "Per me, è una delle domande più grandi in microbiologia:perché questi genomi sono così diversi nel contenuto genetico?" spiega Polz. "Una volta che possiamo definire le popolazioni come unità evolutive, possiamo interpretare le frequenze geniche in queste popolazioni alla luce dei processi evolutivi".
I risultati di Polz e colleghi potrebbero aumentare le stime sulla diversità dei microbi, dice Marx. "Quello che penso sia davvero interessante di questo approccio del gruppo di Martin è che in realtà suggeriscono che la complessità che vediamo è ancora più complessa di quanto gli stiamo dando credito. Potrebbero esserci ancora più tipi che sono ecologicamente importanti là fuori, cose che se fossero piante e animali le chiameremmo specie."
Altri autori del MIT sul documento includono Joseph Elsherbini e Jeff Gore. La ricerca è stata sostenuta, in parte, dalla National Science Foundation e dalla Simons Foundation.
 Acido glicirrizico come farmaco candidato per COVID-19
Acido glicirrizico come farmaco candidato per COVID-19
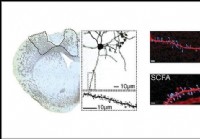 L'integrazione di acidi grassi a catena corta migliora il recupero dall'ictus,
L'integrazione di acidi grassi a catena corta migliora il recupero dall'ictus,
 Sostituire la carne rossa con alternative alla carne a base vegetale riduce il rischio cardiovascolare
Sostituire la carne rossa con alternative alla carne a base vegetale riduce il rischio cardiovascolare
 La vibrazione di tutto il corpo aiuta a ridurre l'infiammazione,
La vibrazione di tutto il corpo aiuta a ridurre l'infiammazione,
 Cambiamento del microbioma respiratorio superiore nei bambini correlato alla suscettibilità alla SARS-CoV-2
Cambiamento del microbioma respiratorio superiore nei bambini correlato alla suscettibilità alla SARS-CoV-2
 Poche prove per la trombocitopenia associata al vaccino mRNA COVID-19,
Poche prove per la trombocitopenia associata al vaccino mRNA COVID-19,
 I probiotici possono offrire benefici terapeutici per i pazienti biopolari
Uno studio del sistema Sheppard Pratt Health di Baltimora ha dimostrato che i probiotici possono fungere da potenziale approccio terapeutico al disturbo bipolare e ad altre condizioni psichiatriche.
I probiotici possono offrire benefici terapeutici per i pazienti biopolari
Uno studio del sistema Sheppard Pratt Health di Baltimora ha dimostrato che i probiotici possono fungere da potenziale approccio terapeutico al disturbo bipolare e ad altre condizioni psichiatriche.
 Gli inibitori di GSK-3 si mostrano promettenti nel trattamento delle infezioni da coronavirus
Ricercatori negli Stati Uniti hanno suggerito un nuovo approccio al trattamento dellinfezione da coronavirus come la sindrome respiratoria acuta grave coronavirus 2 (SARS-CoV-2), lagente che causa la
Gli inibitori di GSK-3 si mostrano promettenti nel trattamento delle infezioni da coronavirus
Ricercatori negli Stati Uniti hanno suggerito un nuovo approccio al trattamento dellinfezione da coronavirus come la sindrome respiratoria acuta grave coronavirus 2 (SARS-CoV-2), lagente che causa la
 Colite ulcerosa
La colite ulcerosa è una malattia che colpisce il colon e il retto. Provoca irritazione e infiammazione e alla fine porta allo sviluppo di ulcere nel rivestimento del colon. La colite ulcerosa è un ti
Colite ulcerosa
La colite ulcerosa è una malattia che colpisce il colon e il retto. Provoca irritazione e infiammazione e alla fine porta allo sviluppo di ulcere nel rivestimento del colon. La colite ulcerosa è un ti