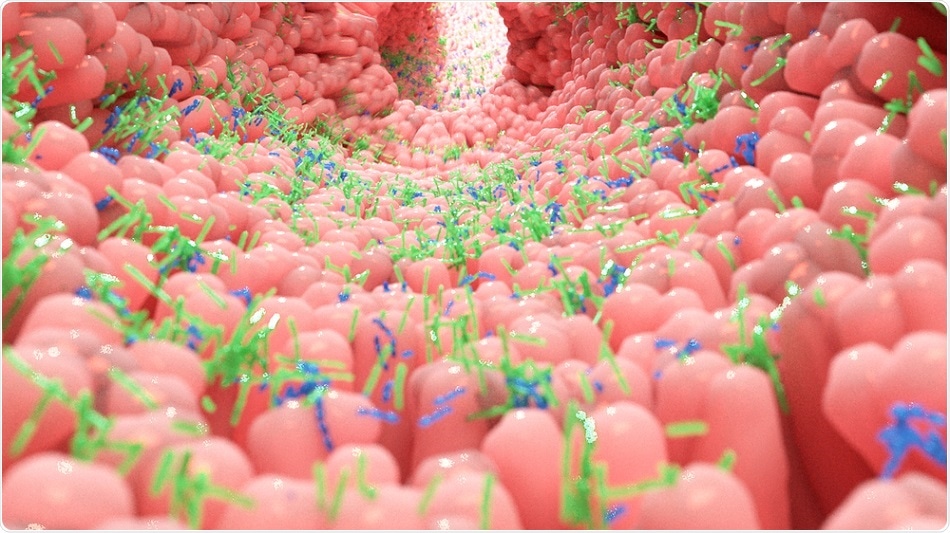
 Alpha Tauri 3D -grafikk | Shutterstock
Alpha Tauri 3D -grafikk | Shutterstock
Studien antydet også at en lignende tilnærming kan brukes til å bestemme dyre- og soppkomponenter i kostholdet vårt.
DNA -strekkoding er en teknikk som bruker korte DNA -sekvenser for raskt å identifisere en organismes art. Over hele kloden, forskere har samarbeidet for å få alle levende ting DNA strekkodet.
DNA -metabarkoding er en spesiell type strekkoding som kan brukes på prøver som inneholder mer enn én organisme. Den bruker de samme referansedatabasene som strekkoding gjør, men tillater taxa fra blandede prøver å bli identifisert ved hjelp av sekvenseringsmetoder med høy gjennomstrømning. Potensialet til teknikken er ekspansivt, Med den kan artssammensetningen bestemmes i praktisk talt alle prøver.
Den nåværende studien, som nylig ble publisert i tidsskriftet mSystemer , viste at kostholds -DNA fra mennesker i avføringsprøver kan forsterkes og sekvenseres ved bruk av teknikker som vanligvis brukes i dyrelivsstudier.
"DNA -sekvensering har gitt oss en stor mengde nye data om ting som mikrobiologi i tarmen og personlig genetikk. Denne studien antyder at den samme kraftige teknologien også kan begynne å fortelle oss om hva vi spiser, som ofte er vanskelig å måle.
Seniorforfatter Lawrence David, fra Duke Center for Genomic and Computational Biology
Mange teknikker er allerede brukt for å vurdere diettinntak, men de fleste av dem er avhengige av folks selvrapportering av hva de har spist. Denne datainnsamlingsmetoden er gjenstand for feil i hukommelsen og skjevhet i rapportering, i tillegg til at den er avhengig av en persons kognitive evne til å svare på undersøkelser.
Ved hjelp av DNA -metabarkoding, forskere kan forsterke DNA som er tilstede i en avføringsprøve og bruke en referansedatabase for å kartlegge sekvensene til matvarer.
"Jeg tenker på DNA -metabarkoding veldig mye som en strekkode på et supermarked. Vi kan tenke på en bestemt DNA -sekvens som en unik identifikator for en bestemt matart, " sier medforfatter Brianna Petrone, også fra Duke University.
David og medforsteforfatter Aspen Reese fra Harvard University, ble bedt om å starte studien etter at de møtte økologene Rob Pringle fra Princeton University og Tyler Kartzinel fra Brown University. Disse forskerne brukte DNA -metabarkoding for å analysere komplekse matveier blant planteetere i den afrikanske savannen.
David og Reese lurte på om metoden også kan brukes til å studere mennesker.
Det er et voksende arbeid i mikrobiomfeltet som indikerer at spesifikke matvarer sannsynligvis vil endre eller forme nivåer av spesifikke bakterier i tarmen, men vi har ofte ikke tilhørende diettdata for mikrobiomstudiene. "
Lawrence David
For studien, forskerne tok DNA ut av kjølelagring som hadde blitt ekstrahert fra avføring brukt i en av deres tidligere studier.
"Vi gjorde tilfeldigvis en studie for et par år siden der vi forberedte mat til deltakere i en mikrobiom diettintervensjon, og vi visste nøyaktig hva de spiste i en gitt uke da avføringen ble samlet inn, " sier David.
De sekvenserte en strekkode -region fra kloroplast -DNA i prøver tatt fra 11 personer som hadde fulgt både fritt utvalgte og kontrollerte dietter.
De forsterket vellykket plante -DNA for omtrent halvparten av prøvene, som økte til 70% når prøver fra personer som bruker et kontrollert, plante-rik diett ble brukt.
Mesteparten av DNA -en som ble sekvensert, matchet matfôrplanter fra mennesker, mye konsumert av mennesker, inkludert korn, grønnsaker og frukt.
"Alt i alt, det var god bred enighet mellom matvarene som ble oppført i dagbøkene som ble ført av studiedeltakerne og de som vi sekvenserte fra avføring, " sier David. "Hvis en mat ble skrevet i diettrekorden, omtrent 80% av tiden, Vi fant det også ved denne metabarkodingsmetoden. "
Den relativt høye PCR -feilen og manglende evne til å skille visse planter på sekvensnivå, gir rom for forbedringer i fremtiden.
For eksempel, kål, brokkoli, kålrabi og rosenkål er alle plantesorter innen samme art, og forskerne klarte ikke å skille mellom dem ved å bruke kloroplast -DNA -sekvensene.
Den eneste maten som ble registrert i dietten som ikke var påvisbar var kaffe, muligens på grunn av at dets DNA har forverret seg eller blitt fortynnet ved steking og brygging.
David forventer at DNA -metabarkoding vil bli brukt i fremtidige studier, så vel som for diettanalyse i tidligere studier.
I likhet med denne studien, Jeg kunne tenke meg at dette ble brukt på arkivert DNA for å se om det er underliggende diettforskjeller som kan forklare noen av mikrobiom -mønstrene som kan ha blitt observert i en studie. Fremover, vi kan også forestille oss at dette blir brukt i nye mikrobiomstudier for å identifisere forhold mellom bestemte matvarer og tarmbakterier, så vel som i bredere studier av ernæring som et supplement til tradisjonelle diettvurderingsteknikker. "
Lawrence David
 At Pepto sannsynligvis ikke vil hjelpe såret ditt
At Pepto sannsynligvis ikke vil hjelpe såret ditt
 Studie avslører antivirale effekter av curcumin
Studie avslører antivirale effekter av curcumin
 DNA -metabarkoding kan forbedre analysen av menneskelig kosthold
DNA -metabarkoding kan forbedre analysen av menneskelig kosthold
 Usunn tarmmikrobiom reduserer synaptisk beskjæring av hjernen,
Usunn tarmmikrobiom reduserer synaptisk beskjæring av hjernen,
 Kan antivirale avledninger av mikroalger bekjempe SARS-CoV-2 og andre virus?
Kan antivirale avledninger av mikroalger bekjempe SARS-CoV-2 og andre virus?
 Flagger potensielle molekylære prediktorer for respons på biologiske terapier ved ulcerøs kolitt
Flagger potensielle molekylære prediktorer for respons på biologiske terapier ved ulcerøs kolitt
 Husholdningsdesinfeksjonsmidler kan bidra til fedme hos barn
Kanadiske forskere har vist at vanlige husholdningsrengjøringsprodukter kan gjøre barn overvektige ved å forårsake endringer i tarmmikrofloraen. Sergey Mironov | Shutterstock Forskerne s
Husholdningsdesinfeksjonsmidler kan bidra til fedme hos barn
Kanadiske forskere har vist at vanlige husholdningsrengjøringsprodukter kan gjøre barn overvektige ved å forårsake endringer i tarmmikrofloraen. Sergey Mironov | Shutterstock Forskerne s
 COVID-19-restriksjoner har ført til 86 prosent fall i norovirusinfeksjoner i USA,
finner studie Utbruddet av COVID-19-pandemien førte til pålegg av flere ikke-farmasøytiske inngrep (NPI) over hele verden. Mens innbyggere i USA har gnidd på råd om å bruke masker offentlig, begrensed
COVID-19-restriksjoner har ført til 86 prosent fall i norovirusinfeksjoner i USA,
finner studie Utbruddet av COVID-19-pandemien førte til pålegg av flere ikke-farmasøytiske inngrep (NPI) over hele verden. Mens innbyggere i USA har gnidd på råd om å bruke masker offentlig, begrensed
 Hvordan øke immunsystemet for å bekjempe koronavirus
Mens verden sliter med koronaviruspandemien, flere og flere mennesker prøver å beskytte seg mot viruset på alle mulige måter. Det er for tiden ingen vaksiner mot alvorlig akutt respiratorisk syndrom c
Hvordan øke immunsystemet for å bekjempe koronavirus
Mens verden sliter med koronaviruspandemien, flere og flere mennesker prøver å beskytte seg mot viruset på alle mulige måter. Det er for tiden ingen vaksiner mot alvorlig akutt respiratorisk syndrom c