La ecología espacial o ecología del paisaje es un área de investigación que investiga la formación de patrones espaciales entre comunidades bacterianas. La magnitud del 'grano' (la unidad más pequeña que se observa) y la 'extensión' (el rango de observaciones) son determinantes importantes de la escala de examen. La escala es crucial cuando se trata de construir teorías sobre cómo se organizan estas comunidades.
¿Cómo se forman esos patrones? En la boca humana por lo menos, la respuesta incluye factores como la temperatura, los niveles de humedad, el flujo de saliva, el nivel de oxígeno, el pH, y el número de veces que se producen abrasiones o procedimientos de higiene bucal. Además de estos factores a nivel macro, los propios microbios producen y utilizan compuestos metabólicos, nutrientes, e inhibidores, incluyendo moléculas antimicrobianas. También evitan físicamente que otros microbios ocupen el espacio principal, o sus superficies pueden ofrecer buenos puntos para que otros microbios se unan. Tales interacciones conducen a una comunidad diversa y funcionalmente redundante, que es más o menos estable y metabólicamente activo según los niveles de interacción inter-microbiana.
Para mapear la orientación espacial, se deben conocer otros factores, como la distancia entre los microbios, así como la distancia entre los microbios y otras características del huésped, como la célula huésped más cercana o la superficie de una biopelícula de la que forma parte el microbio. Las imágenes se utilizan para obtener información sobre dichos patrones a nivel de celda individual a escalas de hasta un milímetro.
El desarrollo de una técnica llamada etiquetado combinatorio junto con la hibridación in situ de fluorescencia de imágenes espectrales (CLASI-FISH) ha ayudado a identificar y localizar múltiples clases microbianas al mismo tiempo al etiquetar cualquier tipo de microbio con múltiples fluoróforos. Esto ayuda a visualizar la disposición espacial de todo un sistema de microbios que forman comunidades microbianas a escala micrométrica.
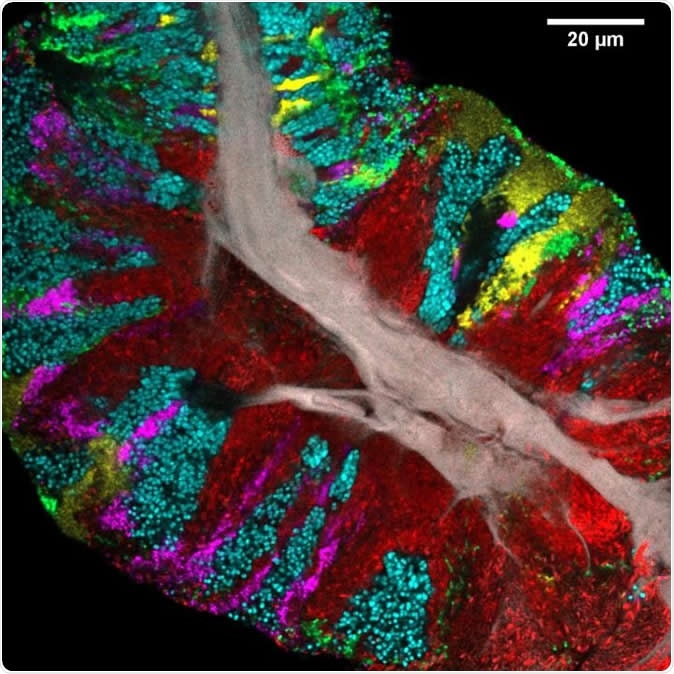 Biopelícula bacteriana raspada de la superficie de la lengua y obtenida con CLASI-FISH. El tejido epitelial humano forma un núcleo central (gris). Los colores indican bacterias diferentes:Actinomyces (rojo) ocupa una región cercana al núcleo; Streptococcus (verde) se localiza en una costra exterior y en rayas en el interior. Otros taxones (Rothia, cian Neisseria, amarillo; Veillonella, magenta) están presentes en grupos y franjas que sugieren el crecimiento de la comunidad hacia afuera desde el núcleo central. Créditos de imagen:Steven Wilbert y Gary Borisy, El Instituto Forsyth
Biopelícula bacteriana raspada de la superficie de la lengua y obtenida con CLASI-FISH. El tejido epitelial humano forma un núcleo central (gris). Los colores indican bacterias diferentes:Actinomyces (rojo) ocupa una región cercana al núcleo; Streptococcus (verde) se localiza en una costra exterior y en rayas en el interior. Otros taxones (Rothia, cian Neisseria, amarillo; Veillonella, magenta) están presentes en grupos y franjas que sugieren el crecimiento de la comunidad hacia afuera desde el núcleo central. Créditos de imagen:Steven Wilbert y Gary Borisy, El Instituto Forsyth El estudio actual utiliza imágenes de fluorescencia multiespectral para establecer su papel en la ecología espacial de los sistemas microbianos de la lengua. Aquí hay múltiples microbios densamente agrupados en contacto con el epitelio humano y también con otros hábitats bucales como la membrana mucosa de la boca y los dientes.
Los investigadores utilizaron un raspador de lengua de plástico estriado para recolectar una muestra raspada de atrás hacia adelante. El tamaño y la disposición interna de estos fragmentos de biopelícula les llevó a concluir que representaban fielmente la disposición espacial de las bacterias en varios niveles del dorso de la lengua en una escala de cientos de micrómetros. Estos niveles incluyen la parte superior de las papilas filiformes que cubren la lengua, los valles entre ellos, y las finas espinas que sobresalen de ellos, todos los cuales hospedan bacterias de diferentes tipos.
Los investigadores identificaron por primera vez los principales tipos de bacterias en las muestras extraídas de las lenguas de 21 voluntarios sanos mediante secuenciación, y luego analizó cada clase para obtener una vista completa de la estructura del microbioma con suficiente detalle para permitir que a cada una de las especies clave se le asigne su propio lugar en la lengua.
La mayoría de los genes microbianos en la comunidad de la lengua están formados por un número limitado de oligotipos, según el Human Microbiome Project (HMP). Al vincular cada oligotipo en la boca a las clases de bacterias en la base de datos ampliada del microbioma oral humano (eHOMD), los investigadores identificaron 17 géneros bacterianos presentes en más del 80% de las personas y que forman el 0,5% de los microbios. Usando datos de secuenciación del HMP, encontraron que el 95% o más de las secuencias bacterianas provenían de un conjunto similar de géneros.
Los investigadores concluyen que es probable que estos géneros "formen el marco espacial y metabólico del microbioma TD sano".
Los investigadores encontraron tres tipos de arreglos microbianos:bacterias libres, bacterias en células epiteliales escamosas, y consorcios bacterianos, o grupos estructuralmente complejos. Estos últimos eran biopelículas bacterianas compuestas por varias capas de microbios, con un límite definido y un núcleo epitelial.
El análisis de la composición bacteriana en cada categoría y ubicación espacial mostró que los consorcios eran más homogéneos que las otras categorías, con patrones bacterianos similares en todas las muestras. Las bacterias libres y unidas al epitelio se produjeron individualmente o en pequeños grupos. A diferencia de, cada consorcio mostró la misma estructura de parche localizada, cada uno dominado por un tipo bacteriano.
Cada parche tiene un límite distinto, tiene decenas a cientos de micrómetros de largo, y tiene un núcleo de células epiteliales de la mucosa humana. Los consorcios viven entre la zona perimetral, que está expuesta a la saliva y al oxígeno, y el núcleo epitelial.
Al menos una muestra de cada participante, y más del 95% de las imágenes de muestra, mostró la presencia de 3 géneros: Actinomyces, Rothia, y Estreptococo . Cada uno tenía su propio 'punto óptimo' en el consorcio, con Actinomyces formando grandes dominios continuos cerca del núcleo o franjas entre parches de otras bacterias. Rothia formaron grandes parches cerca del perímetro, así como alrededor de un núcleo de células epiteliales o bacterianas. Dentro de tal capa cortical, los Rothia a menudo se disgregaba por corrientes o parches de otros tipos bacterianos. Estreptococo formó una fina capa externa en el consorcio, así como venas o parches en su interior.
Otros tipos de bacterias prominentes que se observaron en muestras de todos los individuos del estudio incluyeron Veillonella, Gemella, Neisseriaceae, y phylum Sacaribacterias . Algunas de estas clases de bacterias pueden ayudar a convertir el nitrato en la saliva en nitrito y así ayudar a regular los niveles de óxido nítrico en el cuerpo. Menos de una quinta parte de las células no se tiñeron con ninguna de las sondas específicas.
Próximo, los investigadores analizaron los diferentes géneros dentro del consorcio, todas las especies dentro de un género, y una especie en particular que se pensaba que era la representante de ese género en la lengua. Como se esperaba de los datos de HMP, ellos encontraron, por ejemplo, ese Rothia estuvo representado por R. mucilaginosa , Actinomyces por A. odontolyticus , y en mucha menor medida, A. graevenitizii , y Neisseria por N. flavescens . S. mitis , S. salivarius y S. parasanguinis se encontraron en todos los consorcios pero en diferentes lugares.
Los científicos plantean la hipótesis de que las células bacterianas del dorso de la lengua se empujan unas a otras a medida que se multiplican. Cada clase aumenta más rápidamente en número en el área que es ideal para su crecimiento. dando lugar a parches de forma irregular. Este es el origen de la disposición del parche que se observa en el microbioma maduro. En mala salud, la estructura de la comunidad microbiana puede variar.
"Nuestro estudio es novedoso porque nadie antes había podido observar la biopelícula en la lengua de una manera que distinga todas las bacterias diferentes para que podamos ver cómo se organizan". ", dice el investigador Gary Borisy." La mayor parte del trabajo anterior sobre comunidades bacterianas utilizó enfoques basados en la secuenciación del ADN, pero para obtener la secuencia de ADN, primero tienes que triturar la muestra y extraer el ADN, que destruye toda la hermosa estructura espacial que estaba allí. La obtención de imágenes con nuestra técnica CLASI-FISH nos permite preservar la estructura espacial e identificar las bacterias al mismo tiempo ".
En otras palabras, este método de imágenes pudo identificar la mayoría de las células en cada consorcio, así como su abundancia y disposición espacial en relación con la fuente de nutrición y la ubicación del sustrato.
Los estudios a escala micrométrica ayudan a diferenciar las comunidades microbianas en relación con su disposición biológica. El uso de sondas a nivel de especie muestra que muchas especies son especialistas en sitios que se encuentran en un sitio, como placa dental, pero no en el otro, es decir, el dorso de la lengua.
Los investigadores concluyen, "Aunque las imágenes son solo una de varias tecnologías clave, proporciona el beneficio único de mostrarnos el objetivo:el paisaje y las estructuras que construyen los microbios y que necesitamos explicar y replicar para haber logrado una comprensión de la comunidad microbiana ".
 Las células inmunitarias reparan el intestino dañado en niños con EII
Las células inmunitarias reparan el intestino dañado en niños con EII
 Neil Bell nombrado Director de Desarrollo de Avacta Life Sciences
Neil Bell nombrado Director de Desarrollo de Avacta Life Sciences
 La apoptosis es un mediador importante de la patogénesis en la infección por coronavirus animal
La apoptosis es un mediador importante de la patogénesis en la infección por coronavirus animal
 Ese Pepto probablemente no ayudará a su úlcera
Ese Pepto probablemente no ayudará a su úlcera
 Científico demuestra el papel del microbioma en la obesidad
Científico demuestra el papel del microbioma en la obesidad
 La enfermedad de Parkinson podría prevenirse mediante microbios intestinales
La enfermedad de Parkinson podría prevenirse mediante microbios intestinales
 El microbioma intestinal cambia con la cocción de alimentos vegetales,
dice nuevo estudio Cuando los investigadores de UC San Francisco y Harvard se unieron para descubrir cómo el microbioma intestinal se ve afectado por la cocción, encontraron que ocurren alteraciones p
El microbioma intestinal cambia con la cocción de alimentos vegetales,
dice nuevo estudio Cuando los investigadores de UC San Francisco y Harvard se unieron para descubrir cómo el microbioma intestinal se ve afectado por la cocción, encontraron que ocurren alteraciones p
 Lo que come puede cambiar la forma en que los antibióticos afectan su intestino
Un nuevo estudio realizado por investigadores de la Universidad de Brown en Rhode Island ha descubierto que la dieta puede influir en cómo el tratamiento con antibióticos afecta al microbioma intestin
Lo que come puede cambiar la forma en que los antibióticos afectan su intestino
Un nuevo estudio realizado por investigadores de la Universidad de Brown en Rhode Island ha descubierto que la dieta puede influir en cómo el tratamiento con antibióticos afecta al microbioma intestin
 Pérdida de la barrera epitelial intestinal responsable del MIS-C relacionado con COVID-19 en niños,
sugiere estudiar Si bien la enfermedad por coronavirus 2019 (COVID-19) en niños es relativamente rara y generalmente leve, se sabe que algunos regresan con un raro pero grave, o incluso potencialmente
Pérdida de la barrera epitelial intestinal responsable del MIS-C relacionado con COVID-19 en niños,
sugiere estudiar Si bien la enfermedad por coronavirus 2019 (COVID-19) en niños es relativamente rara y generalmente leve, se sabe que algunos regresan con un raro pero grave, o incluso potencialmente