Raumökologie oder Landschaftsökologie ist ein Forschungsgebiet, das die Bildung räumlicher Muster zwischen Bakteriengemeinschaften untersucht. Die Größe der „Korn“ (die kleinste beobachtete Einheit) und das „Ausmaß“ (der Bereich der Beobachtungen) sind wichtige Determinanten des Untersuchungsmaßstabs. Der Maßstab ist entscheidend, wenn es darum geht, Theorien darüber zu entwickeln, wie solche Gemeinschaften organisiert sind.
Wie entstehen solche Muster? Im menschlichen Mund, wenigstens, die Antwort beinhaltet Faktoren wie Temperatur, der Feuchtigkeitsgehalt, der Speichelfluss, der Sauerstoffgehalt, der pH-Wert, und die Häufigkeit von Abschürfungen oder Mundhygienemaßnahmen. Neben diesen makroökonomischen Faktoren Mikroben selbst produzieren und verwerten Stoffwechselverbindungen, Nährstoffe, und Hemmstoffe, einschließlich antimikrobieller Moleküle. Sie verhindern auch physisch, dass andere Mikroben den Hauptraum einnehmen. oder ihre Oberflächen können anderen Mikroben gute Stellen bieten, um sich daran zu binden. Solche Interaktionen führen zu einer vielfältigen und funktional redundanten Gemeinschaft, die mehr oder weniger stabil und stoffwechselaktiv ist, je nach dem Ausmaß der intermikrobiellen Interaktion.
Um die räumliche Ausrichtung abzubilden, andere Faktoren müssen bekannt sein, wie der Abstand zwischen Mikroben sowie der Abstand zwischen Mikroben und anderen Wirtsmerkmalen wie der nächsten Wirtszelle oder der Oberfläche eines Biofilms, von dem die Mikrobe ein Teil ist. Imaging wird verwendet, um Informationen über solche Muster auf der Ebene einzelner Zellen im Maßstab von bis zu einem Millimeter zu erhalten.
Die Entwicklung einer Technik namens kombinatorische Markierung zusammen mit spektraler Bildgebungs-Fluoreszenz-in-situ-Hybridisierung (CLASI-FISH) hat dazu beigetragen, mehrere Mikrobenklassen gleichzeitig zu identifizieren und zu lokalisieren, indem jede Art von Mikrobe mit mehreren Fluorophoren markiert wurde. Dies hilft, die räumliche Anordnung eines ganzen Systems von Mikroben zu visualisieren, die mikrobielle Gemeinschaften im Mikrometerbereich bilden.
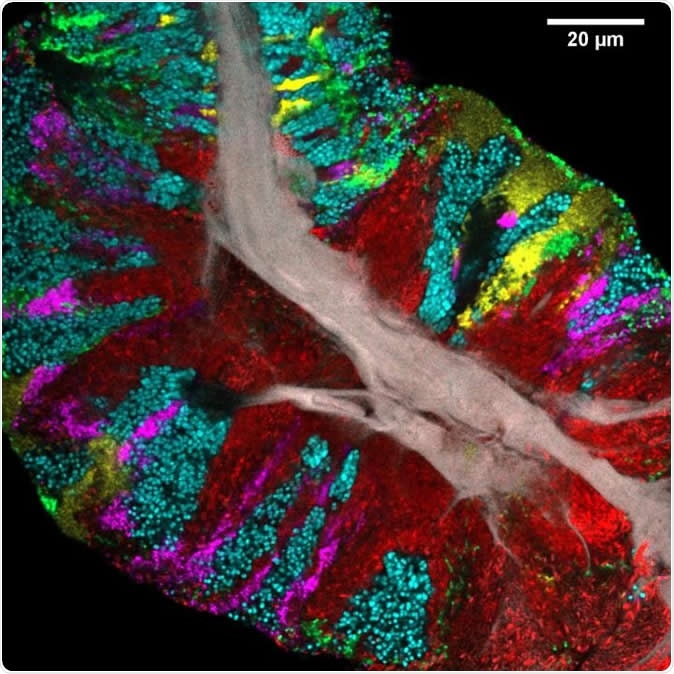 Bakterieller Biofilm, der von der Zungenoberfläche abgekratzt und mit CLASI-FISH abgebildet wurde. Menschliches Epithelgewebe bildet einen zentralen Kern (grau). Farben weisen auf verschiedene Bakterien hin:Actinomyces (rot) besetzen eine kernnahe Region; Streptococcus (grün) ist in einer äußeren Kruste und in Streifen im Inneren lokalisiert. Andere Taxa (Rothia, cyan; Neisseria, Gelb; Veillonella, magenta) sind in Clustern und Streifen vorhanden, die das Wachstum der Gemeinschaft vom zentralen Kern nach außen hindeuten. Bildquelle:Steven Wilbert und Gary Borisy, Das Forsyth-Institut
Bakterieller Biofilm, der von der Zungenoberfläche abgekratzt und mit CLASI-FISH abgebildet wurde. Menschliches Epithelgewebe bildet einen zentralen Kern (grau). Farben weisen auf verschiedene Bakterien hin:Actinomyces (rot) besetzen eine kernnahe Region; Streptococcus (grün) ist in einer äußeren Kruste und in Streifen im Inneren lokalisiert. Andere Taxa (Rothia, cyan; Neisseria, Gelb; Veillonella, magenta) sind in Clustern und Streifen vorhanden, die das Wachstum der Gemeinschaft vom zentralen Kern nach außen hindeuten. Bildquelle:Steven Wilbert und Gary Borisy, Das Forsyth-Institut Die aktuelle Studie verwendet multispektrale Fluoreszenzbildgebung, um ihre Rolle in der räumlichen Ökologie für mikrobielle Systeme auf der Zunge zu ermitteln. Hier befinden sich mehrere dicht gehäufte Mikroben in Kontakt mit dem menschlichen Epithel und auch mit anderen oralen Habitaten wie der Mundschleimhaut und den Zähnen.
Die Forscher verwendeten einen geriffelten Zungenschaber aus Kunststoff, um ein abgekratztes Exemplar von hinten nach vorne zu sammeln. Die Größe und innere Anordnung dieser Biofilmfragmente ließen den Schluss zu, dass sie die räumliche Anordnung der Bakterien auf verschiedenen Ebenen des Zungenrückens über eine Skala von Hunderten von Mikrometern getreu wiedergeben. Diese Ebenen umfassen die Spitzen der fadenförmigen Papillen, die die Zunge bedecken, die Täler zwischen ihnen, und die feinen Stacheln, die aus ihnen herausragen, die alle Bakterien unterschiedlicher Art beherbergen.
Die Forscher identifizierten zuerst die wichtigsten Bakterienarten in den Proben, die von den Zungen von 21 gesunden Freiwilligen durch Sequenzierung abgekratzt wurden. und analysierte dann jede Klasse, um einen vollständigen Überblick über die Struktur des Mikrobioms zu erhalten, der so detailliert war, dass jeder der Schlüsselarten ihre eigene Stelle auf der Zunge zugewiesen werden konnte.
Die meisten mikrobiellen Gene in der Zungengemeinschaft bestehen aus einer begrenzten Anzahl von Oligotypen, nach dem Human Microbiome Project (HMP). Durch die Verknüpfung jedes Oligotyps im Mund mit den Bakterienklassen in der erweiterten Human Oral Microbiome Database (eHOMD) die Forscher identifizierten 17 Bakteriengattungen, die in mehr als 80 % der Menschen vorkommen und 0,5 % der Mikroben bilden. Mithilfe von Sequenzierungsdaten aus dem HMP, Sie fanden heraus, dass 95% oder mehr der bakteriellen Sequenzen aus ähnlichen Gattungen stammten.
Die Forscher kommen zu dem Schluss, dass diese Gattungen "wahrscheinlich sowohl das räumliche als auch das metabolische Gerüst des gesunden TD-Mikrobioms bilden".
Die Forscher fanden drei Arten von mikrobiellen Anordnungen:freie Bakterien, Bakterien auf Plattenepithelzellen, und Bakterienkonsortien, oder strukturell komplexe Gruppen. Letztere waren bakterielle Biofilme aus mehreren Schichten von Mikroben, mit einer deutlichen Grenze und einem epithelialen Kern.
Die Analyse der Bakterienzusammensetzung in jeder Kategorie und räumlichen Lage zeigte, dass Konsortien homogener waren als die anderen Kategorien, mit ähnlichen Bakterienmustern in allen Proben. Freie und epithelgebundene Bakterien traten einzeln oder in kleinen Clustern auf. Im Gegensatz, jedes Konsortium zeigte die gleiche lokalisierte Patch-Struktur, jeweils von einem Bakterientyp dominiert.
Jeder Patch hat eine eigene Grenze, ist Dutzende bis Hunderte von Mikrometern lang, und hat einen Kern aus menschlichen Schleimhautepithelzellen. Die Konsortien leben zwischen der Perimeterzone, die Speichel und Sauerstoff ausgesetzt ist, und der Epithelkern.
Mindestens eine Probe von jedem Teilnehmer, und über 95% der Beispielbilder, zeigte das Vorhandensein von 3 Gattungen: Aktinomyces, Rothia, und Streptokokken . Jeder hatte seinen eigenen "Sweet Spot" im Konsortium, mit Aktinomyces Bildung großer kontinuierlicher Domänen in der Nähe des Kerns oder von Streifen zwischen Flecken anderer Bakterien. Rothia bildeten große Flecken in der Nähe des Umfangs sowie um einen Kern aus Epithel- oder Bakterienzellen. Innerhalb einer solchen kortikalen Schicht das Rothia wurde oft durch Bäche oder Flecken anderer Bakterienarten aufgebrochen. Streptokokken bildete eine dünne äußere Schicht auf dem Konsortium sowie Adern oder Flecken darin.
Andere prominente Bakterienarten, die in Proben von allen Personen in der Studie gefunden wurden, umfassten Veillonella, Gemella, Neisseriaceae, und Stamm Saccharibakterien . Einige dieser Bakterienklassen können dazu beitragen, Nitrat im Speichel in Nitrit umzuwandeln und so den Stickoxidspiegel im Körper zu regulieren. Weniger als ein Fünftel der Zellen wurde mit keiner der spezifischen Sonden gefärbt.
Nächste, die Forscher untersuchten die verschiedenen Gattungen innerhalb des Konsortiums, jede Art innerhalb einer Gattung, und insbesondere eine Art, von der angenommen wurde, dass sie der Vertreter dieser Gattung auf der Zunge ist. Wie von HMP-Daten erwartet, Sie fanden, zum Beispiel, das Rothia wurde vertreten durch R. mucilaginosa , Aktinomyces von A. odontolyticus , und in viel geringerem Maße, A. graevenitizii , und Neisseria von N. flavescens . S. mitis , S. salivarius und S. parasanguinis wurden in allen Konsortien gefunden, jedoch an unterschiedlichen Orten.
Die Wissenschaftler vermuten, dass sich Bakterienzellen auf dem Zungenrücken bei der Vermehrung gegenseitig anstoßen. Jede Klasse nimmt in dem für ihr Wachstum idealen Gebiet schneller zu. führt zu ungleichmäßig geformten Flecken. Dies ist der Ursprung der Patch-Anordnung, die im reifen Mikrobiom zu sehen ist. Bei Krankheit, die Struktur der mikrobiellen Gemeinschaft kann variieren.
„Unsere Studie ist neu, weil noch niemand in der Lage war, den Biofilm auf der Zunge so zu betrachten, dass er all die verschiedenen Bakterien unterscheidet, damit wir sehen können, wie sie sich anordnen. " sagt der Forscher Gary Borisy. "Bei den meisten früheren Arbeiten zu Bakteriengemeinschaften wurden Ansätze auf der Grundlage der aber um die DNA-Sequenz zu erhalten, Sie müssen zuerst die Probe zermahlen und die DNA extrahieren, was all die schöne räumliche Struktur zerstört, die da war. Durch die Bildgebung mit unserer CLASI-FISH-Technik können wir die räumliche Struktur erhalten und gleichzeitig die Bakterien identifizieren."
Mit anderen Worten, diese bildgebende Methode konnte die meisten Zellen in jedem Konsortium identifizieren, sowie deren Häufigkeit und räumliche Anordnung in Bezug auf die Nahrungsquelle und den Standort des Substrats.
Die Studien im Mikrometerbereich helfen, mikrobielle Gemeinschaften in Bezug auf ihre biologische Anordnung zu unterscheiden. Der Einsatz von Sonden auf Artenebene zeigt, dass viele Arten Standortspezialisten sind, die an einem Standort vorkommen, wie Zahnbelag, aber nicht im anderen, das ist, der Zungenrücken.
Die Forscher kommen zu dem Schluss, „Obwohl die Bildgebung nur eine von mehreren Schlüsseltechnologien ist, es bietet den einzigartigen Vorteil, uns das Ziel zu zeigen:die Landschaft und die Strukturen, die Mikroben aufbauen und die wir erklären und replizieren müssen, um ein Verständnis der mikrobiellen Gemeinschaft zu erlangen."
 Bestimmung der säureneutralisierenden Kapazität von OTC-Antazida
Bestimmung der säureneutralisierenden Kapazität von OTC-Antazida
 Forschung sagt bei SARS-CoV-2-Infektion des Hundes,
Forschung sagt bei SARS-CoV-2-Infektion des Hundes,
 Pankreatitis
Pankreatitis
 Proteinbotenstoffe aus dem menschlichen Mikrobiom geben Aufschluss über die menschliche Gesundheit
Proteinbotenstoffe aus dem menschlichen Mikrobiom geben Aufschluss über die menschliche Gesundheit
 Darmstärkende Nahrung kann der Unterernährung bei Kindern weltweit ein Ende setzen
Darmstärkende Nahrung kann der Unterernährung bei Kindern weltweit ein Ende setzen
 Organchip-Technologie verbessert die Untersuchung des Darms für die personalisierte Medizin
Organchip-Technologie verbessert die Untersuchung des Darms für die personalisierte Medizin
 Maßnahmen zur Verhinderung der Übertragung von SARS-CoV-2 durch Abwasser in armen Regionen
Wissenschaftler weltweit bemühen sich zu verstehen, wie sich die Coronavirus-Krankheit 2019 (COVID-19) und der neuartige Erreger, der sie verursacht – das schwere akute respiratorische Syndrom Coronav
Maßnahmen zur Verhinderung der Übertragung von SARS-CoV-2 durch Abwasser in armen Regionen
Wissenschaftler weltweit bemühen sich zu verstehen, wie sich die Coronavirus-Krankheit 2019 (COVID-19) und der neuartige Erreger, der sie verursacht – das schwere akute respiratorische Syndrom Coronav
 Studie mit Zwillingen zeigt, dass COVID-19-Symptome einen genetischen Beitrag haben
Eine neue Studie auf dem Preprint-Server veröffentlicht medRxiv vom Kings College, London, legt nahe, dass die genetische Ausstattung einer Person dazu beitragen kann, wie stark eine Person wahrsche
Studie mit Zwillingen zeigt, dass COVID-19-Symptome einen genetischen Beitrag haben
Eine neue Studie auf dem Preprint-Server veröffentlicht medRxiv vom Kings College, London, legt nahe, dass die genetische Ausstattung einer Person dazu beitragen kann, wie stark eine Person wahrsche
 Frauen, die per Kaiserschnitt geboren wurden, haben ein höheres Risiko für Fettleibigkeit und Diabetes
Eine neue Studie im Open-Access-Journal veröffentlicht JAMA-Netzwerk geöffnet im April 2020 deutet darauf hin, dass Frauen, die per Kaiserschnitt geboren wurden, ein höheres Risiko haben, im Erwachs
Frauen, die per Kaiserschnitt geboren wurden, haben ein höheres Risiko für Fettleibigkeit und Diabetes
Eine neue Studie im Open-Access-Journal veröffentlicht JAMA-Netzwerk geöffnet im April 2020 deutet darauf hin, dass Frauen, die per Kaiserschnitt geboren wurden, ein höheres Risiko haben, im Erwachs