Diese Viren werden Bakteriophagen genannt – wörtlich:Bakterienfresser – kurz Phagen. Phagen gibt es zwar schon lange, diese neuen Phagen verblüffen hinsichtlich ihrer Größe und biologisch komplexen Systeme. Viele ihrer Gene befinden sich normalerweise in Bakterien und werden verwendet, um ihre Wirtsbakterien anzugreifen.
Wo fanden die Wissenschaftler diese seltenen riesigen Phagen? Sie durchforsteten eine riesige Datenbank mit DNA-Sequenzen, die aus einem breiten und vielfältigen Spektrum von Umgebungen auf der Erde stammten. Diese fast 30 Mikrobiome umfassten das Spektrum einer heißen Quelle in Tibet, durch Bioreaktoren in Südafrika, in den Darm einer Frau, die ein Baby im Mutterleib trägt. Andere Orte sind unterirdische Löcher, Ozeane, Seen, und der Darm eines Frühgeborenen.
Die Forscherin Jill Banfield macht dies seit über anderthalb Jahrzehnten. Sie berechnet die Sequenzen aller DNA-Bits in jeder Probe, die sie von einem Ort auf der Erde bekommt. Dann fügt sie die Puzzleteile zusammen, um die genomische Sequenz zu erhalten.
In den meisten Fällen, sie bekommt einen Entwurf des Genoms, bei einigen wurde jedoch bestätigt, dass es sich um das Genom völlig neuer Mikroben handelt. Einige dieser Mikroben werden durch winzige Genome reguliert, die in der Tat, scheinen nicht in der Lage zu sein, das Leben unabhängig zu erhalten, und sind stattdessen vollständig von anderen Lebensformen abhängig, nämlich, Archaeen und Bakterien.
Vor einem Jahr berichtete sie über den Fund großer Phagen, die sie Lak-Phagen nannte, im menschlichen Darm und Mund. Diese Phagen fressen Bakterien im Speichel und im Darm.
Im neuen Papier, sie berichtet über die Isolierung von über 350 sehr großen Phagen mit Genomen, die bei Längen von 200, 000 Basen, viermal größer als die durchschnittliche genetische Sequenz aller bisher bekannten Bakteriophagen (50 kb). Banfield Kommentare, "Wir erforschen das Mikrobiom der Erde, und manchmal tauchen unerwartete Dinge auf."
Das bisher größte sequenzierte Genom umfasst erstaunliche 735, 000 Basenpaare, die etwa 15-mal so groß ist wie das durchschnittliche Phagengenom, Und tatsächlich, deutlich größer als viele Bakteriengenome. Aber es könnten noch größere sein, da sie bisher nur 175 dieser großen Phagen sequenziert haben.
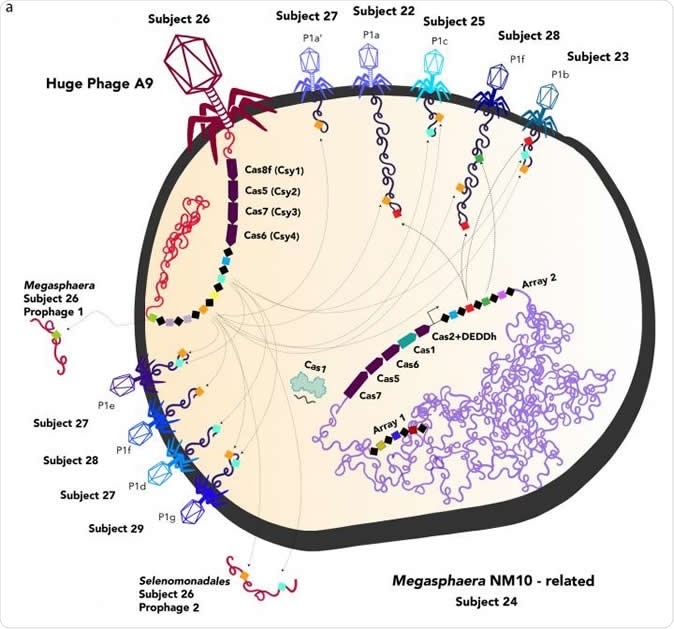 Darstellung riesiger Phagen (rot, links) und normale Phagen, die eine Bakterienzelle infizieren. Der riesige Phagen injiziert seine DNA in die Wirtszelle, wo Cas-Proteine – ein Teil des CRISPR-Immunsystems, das typischerweise nur in Bakterien und Archaeen vorkommt – die Reaktion der Wirtszelle auf andere Viren manipulieren. Das Team der UC Berkeley hat noch keine riesigen Phagen fotografiert, so werden alle dargestellt, die der häufigsten Art von Phagen ähneln, T4. Bildquelle:UC Berkeley Bild mit freundlicher Genehmigung von Jill Banfield lab
Darstellung riesiger Phagen (rot, links) und normale Phagen, die eine Bakterienzelle infizieren. Der riesige Phagen injiziert seine DNA in die Wirtszelle, wo Cas-Proteine – ein Teil des CRISPR-Immunsystems, das typischerweise nur in Bakterien und Archaeen vorkommt – die Reaktion der Wirtszelle auf andere Viren manipulieren. Das Team der UC Berkeley hat noch keine riesigen Phagen fotografiert, so werden alle dargestellt, die der häufigsten Art von Phagen ähneln, T4. Bildquelle:UC Berkeley Bild mit freundlicher Genehmigung von Jill Banfield lab Diese großen Phagen wurden in zehn Kladen oder Gruppen eingeteilt. Jeder wurde "Großer Phage" genannt, obwohl er verschiedene Sprachen verwendet, oder verschiedene Wörter in derselben Sprache. Die Sprachen sind die des Herkunftslandes der Forscher. So gibt es eine Mahaphage-Klade, ein Biggiephage, ein Judaphage, ein Kämpephagen, eine Dakhmphage, ein Jabbarphage und ein Kabirphage, sowie ein Enormephagen, Whopperphage, und Kyodaiphagen-Klade – in Sanskrit, Australisches Englisch, Chinesisch, Dänisch, Arabisch (die nächsten drei), Französisch, Amerikanisches Englisch, und japanisch!
Die bakteriellen Gene in diesen Phagen enthalten einige CRISPR-Elemente, Gen-Editing-Protein kodierende Segmente, die von Bakterien verwendet werden, um viralen Angriffen zu widerstehen. Die Wissenschaftler glauben, dass diese Elemente Teil des CRISPR-Systems des Wirts werden sollen. Dadurch werden die Bakterien befähigt, anderen Viren zu widerstehen, damit diese großen Phagen ihre Mahlzeit ungestört genießen können. Ein anderer Forscher, Basem Al-Shayeb, erklärt, dass diese Phagen dieses System zu ihrem eigenen Vorteil nutzen, "um den Krieg zwischen diesen Viren anzuheizen."
Ein weiteres faszinierendes Element in einem dieser neuartigen Phagen ist ein Protein, das die gleiche Rolle wie das Cas-9-Protein spielt, ein integraler Bestandteil des weit verbreiteten CRISPR-Cas9-Gen-Editing-Tools, das zuerst von Doudna und Charpentier eingeführt wurde. Dieses neue Protein wurde CasØ genannt – der Buchstabe Ø, oder phi (auf Griechisch), ist das Symbol für einen Phagen. Dies ist Teil der Cas-12-Familie, aber diese Phagen haben auch andere bakterielle CRISPR-Elemente wie Cas-9, Cas-X, und Cas-Y-Proteine.
Einige dieser Phagen haben auch sehr große CRISPR-Arrays. Dies bezieht sich auf das Vorhandensein von Arrays, die aus viralen DNA-Fragmenten bestehen, Speicher-Arrays, die die schnelle Erkennung eines wiederholten Angriffs durch einen dieser Viren ermöglichen. Dies, im Gegenzug, löst sofortige Cas-Reaktionen aus, Dies ermöglicht ein spezifisches Targeting dieser Viren.
Ein weiterer überraschender Befund war das Auftreten von Genen, die ribosomale Proteine kodieren. Das Ribosom ist die Zellorganelle, die mRNA liest und daraus spezifische Proteine herstellt. Dies ist eines der ersten Vorkommen von ribosomalen Genen in Viren.
Sie fanden auch Gene, die für Transfer-RNAs kodieren, das sind die Moleküle, die die richtigen Aminosäuren aufnehmen, um in die Proteinsequenz eingebaut zu werden. Es gibt regulatorische Gene für tRNAs, Gene, um den Proteinsyntheseprozess einzuschalten, und sogar ein paar ribosomale Segmente. Diese Gene kommen nur in Lebewesen vor, da sie mit der Herstellung von Proteinbestandteilen zu tun haben – anders als alle anderen Viren können. und das ist eine Fähigkeit, die das Leben buchstabiert.
Ermittler Rohan Sachdeva sagt, dies sei "eines der wichtigsten Merkmale, die Viren und Bakterien voneinander trennen. Nichtleben und Leben." Er nennt dies "die Linie ein bisschen verwischen".
Warum gibt es diese Gene? Die Forscher glauben, dass sie verwendet werden könnten, um die Ribosomen zu entführen, um mit der Replikation viraler Proteine statt bakterieller Wirtsproteine zu beginnen. Ein weiterer Befund ist das Vorhandensein alternativer genetischer Codes, oder die Verwendung von mehr als einem genetischen Code, um dieselbe Aminosäure zu bezeichnen. Diese Eigenschaft könnte das bakterielle Ribosom aus der Bahn werfen und es dazu bringen, die virale RNA zu entschlüsseln und nicht die RNA des Wirts.
Die Entdeckung neuer Werkzeuge im Kampf zwischen Bakterien und Viren bietet viele Möglichkeiten, neue Werkzeuge zur Gen-Editierung zu finden. Viele der neuen Gene müssen noch auf ihre Funktionen untersucht werden, und es könnte gut sein, dass diese neuen Proteine in verschiedenen Anwendungen in der Industrie hilfreich sein werden, die medizinischen Wissenschaften, oder in der Landwirtschaft.
Es gibt Gefahren, auch. Viren übertragen Gene von einem Bakterium auf ein anderes – einige davon könnten für Antibiotikaresistenzen oder für Virulenz (die Fähigkeit, Krankheiten auszulösen) verantwortlich sein. Diese Eigenschaft der neu entdeckten Phagen könnte bedeuten, dass die Gefahr besteht, einige dieser schädlichen Gene in das menschliche Mikrobiom zu übertragen, da Phagen neben Bakterien vorkommen.
Da die Anzahl der Gene, die von diesen großen Phagen getragen werden, deutlich höher ist als die von gewöhnlichen Phagen, sie haben eine größere Fähigkeit, sich um Gene herum zu bewegen, die andere Zellen verletzen können. Im Gegenzug, dies erhöht das Risiko, dass einige dieser Gene von Genen in der menschlichen Umwelt übernommen werden.
Insgesamt, Die Forscher sind fasziniert von der Präsenz von Phagen mit riesigen Genomen in einer Vielzahl von Mikrobiomen auf der ganzen Erde. Die Verwandtschaft zwischen diesen großen Phagen wird so interpretiert, dass sie einer alten Familie von Viren mit großem Genom entstammen.
Sagt Banfield, „Große Genome zu haben, ist eine erfolgreiche Existenzstrategie. Diese Bakterienviren sind ein Teil der Biologie, von replizierenden Einheiten, über die wir sehr wenig wissen."
Wahr genug. Diese Phagen haben eine Größe, die sie irgendwo in die Lücke zwischen den Archaeen (den frühesten Verwandten der Bakterien), und gewöhnliche Phagen, die wie nicht lebende Dinge aussehen. Banfield beschreibt sie als „erfolgreiche Existenzstrategien, die Hybriden zwischen dem sind, was wir als traditionelle Viren und traditionelle lebende Organismen bezeichnen“.
 Studie deutet auf Zusammenhang zwischen probiotischem Konsum und „Gehirnnebel“ hin
Studie deutet auf Zusammenhang zwischen probiotischem Konsum und „Gehirnnebel“ hin
 Probiotika können in den nächsten zwei Jahrzehnten helfen, Mangelernährung einzudämmen.
Probiotika können in den nächsten zwei Jahrzehnten helfen, Mangelernährung einzudämmen.
 Probiotika als adjuvante Therapie für COVID-19-Patienten
Probiotika als adjuvante Therapie für COVID-19-Patienten
 Das Darmmikrobiom ist auch im fetalen Leben Realität
Das Darmmikrobiom ist auch im fetalen Leben Realität
 Die langfristige Anwendung von Antibiotika bei Frühgeborenen fördert arzneimittelresistente Darmbakterien
Die langfristige Anwendung von Antibiotika bei Frühgeborenen fördert arzneimittelresistente Darmbakterien
 Wissenschaftler entwickeln Peptide, die das Gleichgewicht der Darmbakterien wiederherstellen und Arteriosklerose umkehren
Wissenschaftler entwickeln Peptide, die das Gleichgewicht der Darmbakterien wiederherstellen und Arteriosklerose umkehren
 Uralte Primaten-Mikrobiome könnten mehr Informationen über die menschliche Entwicklung liefern
Uralte menschliche Mikrobiome sind unter dem Mikroskop für das, was sie Wissenschaftlern über die Menschen von vor langer Zeit erzählen. Eine neue Studie in der Zeitschrift veröffentlicht Grenzen in
Uralte Primaten-Mikrobiome könnten mehr Informationen über die menschliche Entwicklung liefern
Uralte menschliche Mikrobiome sind unter dem Mikroskop für das, was sie Wissenschaftlern über die Menschen von vor langer Zeit erzählen. Eine neue Studie in der Zeitschrift veröffentlicht Grenzen in
 Gentechnisch veränderte Darmbakterien reduzieren das Risiko von Darmkrebs bei Mäusen, findet Studie
Forscher haben herausgefunden, dass die Bearbeitung der Gene der Bakterien im Darm von Mäusen dazu beitragen könnte, die Entzündung und das damit verbundene Risiko von Darmkrebs zu reduzieren. Die For
Gentechnisch veränderte Darmbakterien reduzieren das Risiko von Darmkrebs bei Mäusen, findet Studie
Forscher haben herausgefunden, dass die Bearbeitung der Gene der Bakterien im Darm von Mäusen dazu beitragen könnte, die Entzündung und das damit verbundene Risiko von Darmkrebs zu reduzieren. Die For
 Neil Bell zum Chief Development Officer von Avacta Life Sciences ernannt
Avacta-Gruppe plc, der Entwickler von Affimer ® Biotherapeutika und Reagenzien, freut sich, die Ernennung von Neil Bell zum Chief Development Officer von Avacta Life Sciences mit sofortiger Wirkung
Neil Bell zum Chief Development Officer von Avacta Life Sciences ernannt
Avacta-Gruppe plc, der Entwickler von Affimer ® Biotherapeutika und Reagenzien, freut sich, die Ernennung von Neil Bell zum Chief Development Officer von Avacta Life Sciences mit sofortiger Wirkung