Disse vira kaldes bakteriofager - bogstaveligt talt, spisere af bakterier - og fag for kort. Mens fager har eksisteret i lang tid, disse nye fager er overraskende med hensyn til deres størrelse og biologisk komplekse systemer. Mange af deres gener findes normalt i bakterier og bruges til at angribe deres værtsbakterier.
Hvor fandt forskerne disse sjældne enorme fager? De undersøgte en stor database med DNA -sekvenser hentet fra en bred og forskelligartet række miljøer på jorden. Disse næsten 30 mikrobiomer strakte sig over spektret fra en varm kilde i Tibet, gennem bioreaktorer i Sydafrika, til tarmen på en kvinde, der bar en baby i livmoderen. Andre steder omfatter underjordiske huller, oceaner, søer, og tarmen hos en præmatur baby.
Det har forsker Jill Banfield gjort i over halvandet årti. Hun beregner sekvenserne af alle DNA -bitene i enhver prøve, hun får fra et sted på jorden. Hun passer derefter puslespilsbrikkerne sammen for at få den genomiske sekvens.
I de fleste tilfælde, hun får et udkast til genomet, men nogle få er blevet bekræftet at være genomer for helt nye mikrober. Nogle af disse mikrober er reguleret af små genomer, som, Ja, synes ude af stand til at opretholde livet uafhængigt, og i stedet er helt afhængige af andre livsformer, nemlig, archaea og bakterier.
Det var et år siden, at hun rapporterede fundet af store fag, som hun kaldte Lak -fager, i menneskets tarm og mund. Disse fager spiser bakterier i spyt og tarm.
I det nye papir, hun rapporterer isolering af over 350 meget store fager med genomer, der er, i længder på 200, 000 baser, fire gange større end den gennemsnitlige genetiske sekvens for enhver hidtil kendt bakteriofag (50 kb). Banfield kommentarer, "Vi udforsker Jordens mikrobiomer, og nogle gange dukker der uventede ting op. "
Den største sekvenserede hidtil har et genom, der spænder over en forbløffende 735, 000 basepar, hvilket er omkring 15 gange størrelsen af det gennemsnitlige faggenom, og faktisk, markant større end mange bakterielle genomer. Men der kan stadig være større, da de kun har sekventeret 175 af disse store fag indtil nu.
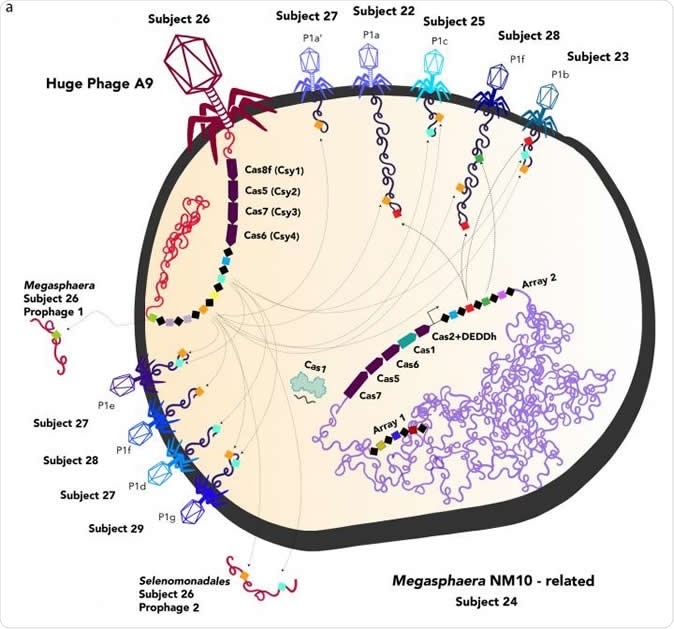 Skildring af enorme fager (rød, til venstre) og normale fager, der inficerer en bakteriecelle. Den enorme fag injicerer sit DNA i værtscellen, hvor Cas -proteiner - en del af CRISPR -immunsystemet typisk kun findes i bakterier og archaea - manipulerer værtscellens reaktion på andre vira. UC Berkeley -teamet har endnu ikke fotograferet nogen store fager, så alle er afbildet ligner den mest almindelige type fag, T4. Billedkredit:UC Berkeley billede med tilladelse fra Jill Banfield lab
Skildring af enorme fager (rød, til venstre) og normale fager, der inficerer en bakteriecelle. Den enorme fag injicerer sit DNA i værtscellen, hvor Cas -proteiner - en del af CRISPR -immunsystemet typisk kun findes i bakterier og archaea - manipulerer værtscellens reaktion på andre vira. UC Berkeley -teamet har endnu ikke fotograferet nogen store fager, så alle er afbildet ligner den mest almindelige type fag, T4. Billedkredit:UC Berkeley billede med tilladelse fra Jill Banfield lab Disse store fager blev klassificeret i ti klader eller grupper. Hver har fået navnet "Big fag", selvom de bruger forskellige sprog, eller forskellige ord på samme sprog. Sprogene er forskernes oprindelsesland. Således er der en Mahaphage -klade, en Biggiephage, en Judaphage, en Kaempephage, en Dakhmphage, en Jabbarphage og en Kabirphage, samt en Enormephage, Whopperphage, og Kyodaiphage -klade - på sanskrit, Australsk engelsk, Kinesisk, Dansk, Arabisk (de næste tre), Fransk, Amerikansk engelsk, og japansk!
Bakteriegenerne i disse fager indeholder nogle CRISPR -elementer, genredigerende proteinkodende segmenter, der bruges af bakterier til at modstå virusangreb. Forskerne mener, at disse elementer er beregnet til at blive en del af værtens CRISPR -system, give bakterierne mulighed for at modstå andre vira, så disse store fager kan nyde deres måltid uafbrudt. En anden forsker, Basem Al-Shayeb, forklarer, at disse fager bruger dette system til egen fordel, "at brænde krig mellem disse vira."
Et andet fascinerende element i en af disse nye fager er et protein, der spiller den samme rolle som Cas-9-proteinet, en integreret del af det meget udbredte CRISPR-Cas9 genredigeringsværktøj, der først blev introduceret af Doudna og Charpentier. Dette nye protein er blevet kaldt CasØ - bogstavet Ø, eller phi (på græsk), er symbolet for en fag. Dette er en del af Cas-12 familien, men disse fager har også andre bakterielle CRISPR-elementer som Cas-9, Cas-X, og Cas-Y proteiner.
Nogle af disse fager har også meget store CRISPR -arrays. Dette refererer til tilstedeværelsen af arrays sammensat af virale DNA -fragmenter, hukommelsesarrays, der muliggør hurtig genkendelse af et gentaget angreb af nogen af disse vira. Det her, på tur, udløser øjeblikkelige Cas -reaktioner, muliggør specifik målretning af disse vira.
Et andet opsigtsvækkende fund var forekomsten af gener, der koder for ribosomale proteiner. Ribosomet er celleorganellen, der læser mRNA og bruger det til at lave specifikke proteiner. Dette er blandt de første forekomster af ribosomale gener i vira.
De fandt også gener, der koder for overførsel af RNA'er, som er molekylerne, der opfanger de rigtige aminosyrer, der skal inkorporeres i proteinsekvensen. Der er regulatoriske gener for tRNA'er, gener til at tænde proteinsynteseprocessen, og endda et par ribosomale segmenter. Disse gener findes kun i livsformer, da de har at gøre med fremstilling af proteinkomponenter - hvilket er ulig hvad enhver anden virus kan, og som er en evne, der staver liv.
Efterforsker Rohan Sachdeva siger, at dette er "en af de vigtigste definerende funktioner, der adskiller virus og bakterier, ikke-liv og liv. "Han kalder dette" at sløre linjen lidt. "
Hvorfor er disse gener der? Forskerne tror, at de kan bruges til at kapre ribosomerne for at begynde at replikere virale frem for at være vært for bakterielle proteiner. Et yderligere fund er tilstedeværelsen af alternative genetiske koder, eller brug af mere end én genetisk kode til at betegne den samme aminosyre. Denne funktion kunne godt smide det bakterielle ribosom af sporet og narre det til at afkode det virale RNA frem for værtens eget RNA.
Opdagelsen af nye værktøjer, der bruges i kampen mellem bakterier og vira, giver mange muligheder for at finde nye genredigeringsværktøjer. Mange af de nye gener mangler stadig at blive undersøgt for deres funktioner, og det kan godt være, at disse nye proteiner vil være nyttige i forskellige applikationer i industrien, lægevidenskaben, eller i landbruget.
Der er farer, også. Virus overfører gener fra en bakterie til en anden - hvoraf nogle kan være ansvarlige for antibiotikaresistens eller for virulens (evnen til at forårsage sygdom). Denne egenskab ved de nyopdagede fager kan betyde, at der er risiko for at overføre nogle af disse skadelige gener til det humane mikrobiom, da fager forekommer sammen med bakterier.
Da antallet af gener båret af disse store fager er markant højere end almindelige fager, de har en større evne til at bevæge sig rundt om gener, der kan skade andre celler. På tur, dette øger risikoen for, at nogle af disse gener vil blive erhvervet af gener i det menneskelige miljø.
Alt i alt, forskerne er fascineret af tilstedeværelsen af fager med enorme genomer i en række mikrobiomer over hele jorden. Forholdet mellem disse store fager fortolkes som, at de stammer fra en gammel familie af store genomvira.
Siger Banfield, "At have store genomer er en vellykket eksistensstrategi. Disse vira af bakterier er en del af biologien, af replikerende enheder, som vi ved meget lidt om. ”
Sandt nok. Disse fager er af en størrelse, der får dem til at passe et sted i kløften mellem archaea (de tidligste slægtninge til bakterier), og almindelige fager, der ligner ikke-levende ting. Banfield beskriver dem som "vellykkede eksistensstrategier, der er hybrider mellem det, vi tænker på som traditionelle vira og traditionelle levende organismer."
 Hvorfor har jeg brug for en koloskopi?
Hvorfor har jeg brug for en koloskopi?
 Proteinsendinger fra humant mikrobiom kaster lys over menneskers sundhed
Proteinsendinger fra humant mikrobiom kaster lys over menneskers sundhed
 Typen af øvre luftvejsbakterier kan påvirke astmaens sværhedsgrad
Typen af øvre luftvejsbakterier kan påvirke astmaens sværhedsgrad
 Mennesker kontra vira - Kan vi undgå udryddelse i nær fremtid?
Mennesker kontra vira - Kan vi undgå udryddelse i nær fremtid?
 Opdagelse af 100 nye gener kan hjælpe forskning i pigmenteringssygdomme
Opdagelse af 100 nye gener kan hjælpe forskning i pigmenteringssygdomme
 Ulcerøs colitis
Ulcerøs colitis
 Oral sex kan udløse bakteriel vaginose
En ny undersøgelse offentliggjort i tidsskriftet PLOS Biologi har afsløret forskellige mikrobielle populationer kan lette vaginal kolonisering af patogener, som igen kan forlænge egenskaberne ved ba
Oral sex kan udløse bakteriel vaginose
En ny undersøgelse offentliggjort i tidsskriftet PLOS Biologi har afsløret forskellige mikrobielle populationer kan lette vaginal kolonisering af patogener, som igen kan forlænge egenskaberne ved ba
 Hvide blodlegemer og deres rolle i hjernen
I et banebrydende studie, en gruppe internationale forskere har fundet ud af, at hjernen har specifikke interne immunceller, der hjælper med normal hjernens udvikling og spiller en rolle i visse neuro
Hvide blodlegemer og deres rolle i hjernen
I et banebrydende studie, en gruppe internationale forskere har fundet ud af, at hjernen har specifikke interne immunceller, der hjælper med normal hjernens udvikling og spiller en rolle i visse neuro
 Hvorfor COVID-19-patienter har flere patogene bakterier i næsen
Forskere sammenlignede nasal mikrobiomet hos patienter med coronavirus sygdom 2019 (COVID-19), raske individer, og sundhedspersonale. Disse undersøgelser indikerede en stigning i patogenet Pseudomona
Hvorfor COVID-19-patienter har flere patogene bakterier i næsen
Forskere sammenlignede nasal mikrobiomet hos patienter med coronavirus sygdom 2019 (COVID-19), raske individer, og sundhedspersonale. Disse undersøgelser indikerede en stigning i patogenet Pseudomona