La recherche est publiée dans le Actes de l'Académie nationale des sciences .
La biologie structurale a été capable de résoudre la structure des virus avec une résolution étonnante, jusqu'à chaque atome de chaque protéine. Mais nous ne savions toujours pas comment cette structure s'assemble. Notre technique donne la première fenêtre sur la façon dont les virus s'assemblent et révèle la cinétique et les voies en détail quantitatif. »
Vinothan Manoharan, la famille Wagner, professeur de génie chimique et professeur de physique à la Harvard John A. Paulson School of Engineering and Applied Sciences
Manoharan est également co-directeur de la Quantitative Biology Initiative, un effort inter-Harvard qui rassemble la biologie, nouvelles techniques de mesure, statistiques et mathématiques pour développer la causalité, modèles mathématiques prédictifs des systèmes biologiques.
Manoharan et son équipe se sont concentrés sur les virus à ARN simple brin, le type de virus le plus abondant sur la planète. Chez l'homme, Les virus à ARN sont responsables, entre autres, fièvre du Nil occidental, gastro-entérite, main, pied, et la maladie de la bouche, polio, et le rhume.
Ces virus ont tendance à être très simples. Le virus que Manoharan et son équipe ont étudié, qui infecte la bactérie E. coli, est d'environ 30 nanomètres de diamètre et a un morceau d'ARN, avec environ 3600 nucléotides, et 180 protéines identiques. Les protéines s'organisent en hexagones et pentagones pour former une structure semblable à un ballon de football autour de l'ARN, appelé capside.
Comment ces protéines parviennent à former cette structure est la question centrale dans l'assemblage du virus. Jusqu'à maintenant, personne n'avait pu observer l'assemblage viral en temps réel car les virus et leurs composants sont très petits et leurs interactions sont très faibles.
Pour observer les virus, les chercheurs ont utilisé une technique optique connue sous le nom de microscopie à diffusion interférométrique, dans lequel la lumière diffusée par un objet crée une tache sombre dans un champ de lumière plus large. La technique ne révèle pas la structure du virus mais elle révèle sa taille et comment cette taille change avec le temps.
Les chercheurs ont attaché des brins d'ARN viral à un substrat, comme les tiges d'une fleur, et des protéines coulaient à la surface. Puis, à l'aide du microscope interférométrique, ils ont vu des taches sombres apparaître et devenir de plus en plus sombres jusqu'à ce qu'elles aient la taille de virus adultes. En enregistrant les intensités de ces taches de croissance, les chercheurs ont pu déterminer combien de protéines se sont attachées à chaque brin d'ARN au fil du temps.
"Une chose que nous avons remarquée immédiatement, c'est que l'intensité de tous les points a commencé faible, puis a atteint l'intensité d'un virus complet, " Manoharan a déclaré. " Cette fusillade s'est produite à des moments différents. Quelques capsides assemblées en moins d'une minute, certains en ont pris deux ou trois, et certains en ont pris plus de cinq. Mais une fois qu'ils ont commencé à s'assembler, ils n'ont pas fait marche arrière. Ils ont grandi et grandi et puis ils ont fini. »
Les chercheurs ont comparé ces observations aux résultats antérieurs de simulations, qui a prédit deux types de voies d'assemblage. Dans un type de parcours, les protéines se collent d'abord au hasard à l'ARN puis se réarrangent en une capside. Dans la seconde, une masse critique de protéines, appelé noyau, doit se former avant que la capside puisse se développer.
Les résultats expérimentaux correspondaient à la deuxième voie et excluaient la première. Le noyau se forme à des moments différents pour différents virus, mais une fois qu'il le fait, le virus se développe rapidement et ne s'arrête pas jusqu'à ce qu'il atteigne sa bonne taille.
Les chercheurs ont également remarqué que les virus avaient tendance à se désassembler plus souvent lorsqu'il y avait plus de protéines circulant sur le substrat.
"Les virus qui s'assemblent de cette manière doivent équilibrer la formation de noyaux avec la croissance de la capside. Si les noyaux se forment trop rapidement, les capsides complètes ne peuvent pas se développer. Cette observation pourrait nous donner quelques indications sur la façon de faire dérailler l'assemblage de virus pathogènes, " dit Manoharan.
Comment les protéines individuelles se réunissent pour former le noyau est encore une question ouverte, mais maintenant que les expérimentateurs ont identifié la voie, les chercheurs peuvent développer de nouveaux modèles qui explorent l'assemblage au sein de cette voie. Ces modèles pourraient également être utiles pour concevoir des nanomatériaux qui s'assemblent eux-mêmes.
"C'est un bon exemple de biologie quantitative, en ce que nous avons des résultats expérimentaux qui peuvent être décrits par un modèle mathématique, " dit Manoharan.
 Le microbiome humain coupe les glycanes muqueux,
Le microbiome humain coupe les glycanes muqueux,
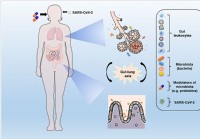 La modulation du microbiote et le rétablissement de l'eubiose pourraient aider à freiner les complications du COVID-19
La modulation du microbiote et le rétablissement de l'eubiose pourraient aider à freiner les complications du COVID-19
 Produit chimique contre le cancer provenant d'un microbe intestinal commun
Produit chimique contre le cancer provenant d'un microbe intestinal commun
 Neil Bell nommé directeur du développement d'Avacta Life Sciences
Neil Bell nommé directeur du développement d'Avacta Life Sciences
 Les bactéries lactiques et les bactéries intestinales contribuent aux bienfaits du seigle sur la santé,
Les bactéries lactiques et les bactéries intestinales contribuent aux bienfaits du seigle sur la santé,
 Allergies liées à une consommation plus élevée de malbouffe,
Allergies liées à une consommation plus élevée de malbouffe,
 Le microbiote rural et urbain diffère dès le plus jeune âge,
trouve une nouvelle étude Une nouvelle étude a montré que les différences dans le microbiome intestinal humain entre les personnes vivant dans les zones rurales et urbaines du Nigeria commencent à un
Le microbiote rural et urbain diffère dès le plus jeune âge,
trouve une nouvelle étude Une nouvelle étude a montré que les différences dans le microbiome intestinal humain entre les personnes vivant dans les zones rurales et urbaines du Nigeria commencent à un
 Les scientifiques développent des peptides qui rétablissent l'équilibre des bactéries intestinales et inversent l'athérosclérose
Les chercheurs continuent dexplorer le rôle des bactéries intestinales sur la santé, y compris son lien avec la dépression, maladies auto-immunes, santé mentale, et lobésité. Maintenant, une équipe
Les scientifiques développent des peptides qui rétablissent l'équilibre des bactéries intestinales et inversent l'athérosclérose
Les chercheurs continuent dexplorer le rôle des bactéries intestinales sur la santé, y compris son lien avec la dépression, maladies auto-immunes, santé mentale, et lobésité. Maintenant, une équipe
 Selon une étude, les produits de nettoyage peuvent augmenter le risque d'asthme chez les enfants
Selon une nouvelle étude de chercheurs de la Faculté des sciences de la santé (SFU) de lUniversité Simon Fraser, lexposition aux produits dentretien ménager pendant que les bébés pourraient augmenter
Selon une étude, les produits de nettoyage peuvent augmenter le risque d'asthme chez les enfants
Selon une nouvelle étude de chercheurs de la Faculté des sciences de la santé (SFU) de lUniversité Simon Fraser, lexposition aux produits dentretien ménager pendant que les bébés pourraient augmenter