p A questão emocionante é se esses são os patógenos de Braak capazes de desencadear a DP, ou são irrelevantes para DP, mas capazes de penetrar no intestino e crescer, porque o revestimento do intestino está comprometido na DP. Ressaltamos que nenhuma reclamação pode ser feita em função baseada unicamente em associação. A identidade desses microrganismos permitirá estudos experimentais para determinar se e como eles desempenham um papel na DP. "p Payami e colegas da UAB, Emory University, O Albany Medical College e a University of Washington foram capazes de identificar esses microrganismos porque realizaram o maior estudo de associação do microbioma de pessoas com DP e controles até o momento. Muitos estudos anteriores encontraram microbiomas intestinais alterados em pessoas com DP, mas não detectaram um aumento nos patógenos oportunistas. Patógenos oportunistas muitas vezes são inofensivos, mas podem crescer e causar infecções se o sistema imunológico estiver comprometido ou se penetrarem em locais estéreis do corpo. p "Suspeitamos que a razão pela qual fomos capazes de detectar esses microrganismos é que eles são raros e tínhamos uma amostra e poder muito maiores do que os estudos anteriores, "Payami disse. Seus pesquisadores reanalisaram seu estudo de 2017, que tinha 197 casos e 130 controles, usando um pipeline de bioinformática mais avançado. Eles também analisaram um novo, conjunto de dados independente com 323 casos de PD e 184 controles, em paralelo ao primeiro conjunto de dados. Isso permitiu a replicação interna e o poder de detectar sinais raros e comuns. Os estudos anteriores do microbioma de DP variaram de 10 a 197 casos de DP e de 10 a 130 controles. p Um estudo de associação de todo o microbioma usa avanços no sequenciamento de DNA e ferramentas computacionais para procurar comunidades microbianas que podem estar associadas a doenças. Está surgindo a compreensão de que o microbioma intestinal - que inclui 500 para 1, 000 espécies bacterianas que têm uma influência principalmente benéfica - desempenham um papel importante na saúde e nas doenças humanas. p Payami e colegas também usaram a análise de rede de correlação livre de hipóteses para identificar comunidades de microorganismos co-ocorrentes. A análise de rede é uma nova ferramenta importante em biologia. Um exemplo de rede de fácil compreensão é uma rede social como o Facebook, onde se pode mapear as conexões entre seguidores ou amigos. Algumas pessoas terão um grande número de conexões, alguns terão muitos, e a grande maioria terá muito menos. Um mapa dessas conexões é semelhante a um mapa de rota de uma companhia aérea. p Usando análise de rede, Payami e colegas encontraram três aglomerados polimicrobianos, e também descobriu que cada cluster compartilhava características funcionais. O primeiro grupo era de patógenos oportunistas superabundantes em casos de DP, um novo achado. p Os outros dois clusters foram uma confirmação de estudos anteriores. Em comparação com os controles, pessoas com DP apresentaram níveis reduzidos de um grupo de micróbios que produzem ácidos graxos de cadeia curta. No terceiro cluster, as pessoas com DP tinham níveis elevados de dois gêneros que são micróbios probióticos que metabolizam carboidratos. p Payami diz que o estudo atual teve um foco preciso e uma execução analítica intencionalmente estrita. O rigor do estudo incluiu mostrar que os microbiomas intestinais alterados nos casos de DP eram independentes do sexo, era, IMC, constipação, desconforto gastrointestinal, geografia e dieta. Os 15 gêneros associados a PD que alcançaram significância em todo o microbioma em ambos os conjuntos de dados foram identificados usando dois métodos, e com ou sem ajuste de covariável. p "Há mais a ser aprendido, "Payami disse, "com tamanhos de amostra maiores com maior poder, estudos longitudinais para rastrear a mudança de doença prodrômica para doença avançada, e por sequenciamento de metagenoma de próxima geração para ampliar o escopo de bactérias e arquéias para incluir vírus e fungos, e melhorar a resolução para cepa e nível de gene. " p Coautores com Payami para o estudo, "Caracterizando disbiose do microbioma intestinal em DP:evidências para superabundância de patógenos oportunistas, "publicado no Revista Nature Partner Parkinson's Disease são Zachary D. Wallen, Mary Appah, Marissa N. Dean, Cheryl L. Sesler e David G. Standaert, Departamento de Neurologia da UAB; Stewart A. Factor, Escola de Medicina da Emory University, Atlanta, Geórgia; Eric Molho, Albany Medical College, Albany, Nova york; e Cyrus P. Zabetian, Veterans Administration Puget Sound Health Care System e University of Washington, Seattle, Washington. p O apoio veio do National Institutes of Health concede NS036960, NS062684, NS108675 e NS095775. p A Atividade de Aquisição de Pesquisa Médica do Exército dos EUA, 820 Chandler Street, Fort Detrick, Maryland 21702-5014, é o escritório de aquisição de concessão e administração. Este trabalho também foi apoiado pelo Comando de Material de Pesquisa Médica do Exército dos EUA, endossado pelo Exército dos EUA, através do Programa de Pesquisa de Parkinson, sob os prêmios nos. W81XWH1810508 e W81XWH1810509. Opiniões, interpretações, as conclusões e recomendações são do autor e não são necessariamente endossadas pelo Exército dos EUA. p Ao conduzir pesquisas com animais, o (s) investigador (es) cumprem as leis dos Estados Unidos e os regulamentos do Departamento de Agricultura. Na condução de pesquisas utilizando DNA recombinante, o investigador aderiu às Diretrizes do NIH para pesquisa envolvendo moléculas de DNA recombinante. Na condução de pesquisas envolvendo organismos perigosos ou toxinas, o investigador aderiu ao Guia de Biossegurança em Laboratórios Microbiológicos e Biomédicos do CDC-NIH. p Na UAB, Payami detém a cadeira dotada de neurologia John T. e Juanelle D. Strain.Haydeh Payami, Ph.D., Professor de Neurologia, Universidade do Alabama em Birmingham
 A perda de microbioma devido ao uso de antibióticos afeta a resposta à vacina contra a gripe
A perda de microbioma devido ao uso de antibióticos afeta a resposta à vacina contra a gripe
 Os cientistas transformam o sangue tipo A em tipo O universal,
Os cientistas transformam o sangue tipo A em tipo O universal,
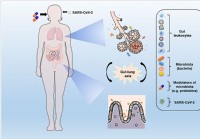 A modulação da microbiota e o restabelecimento da eubiose podem ajudar a conter as complicações do COVID-19
A modulação da microbiota e o restabelecimento da eubiose podem ajudar a conter as complicações do COVID-19
 Fórmula à base de leite de cabra boa para a saúde intestinal de bebês
Fórmula à base de leite de cabra boa para a saúde intestinal de bebês
 Lidando com a síndrome do intestino irritável
Lidando com a síndrome do intestino irritável
 Rotavírus desempenha um papel no desenvolvimento de diabetes tipo 1
Rotavírus desempenha um papel no desenvolvimento de diabetes tipo 1
 Os antioxidantes na dieta podem aumentar o risco de câncer de intestino,
novo estudo revela Os benefícios para a saúde dos antioxidantes nos alimentos foram comprovados em um corpo considerável de literatura científica. Agora, um novo estudo mostra que muito de uma coisa b
Os antioxidantes na dieta podem aumentar o risco de câncer de intestino,
novo estudo revela Os benefícios para a saúde dos antioxidantes nos alimentos foram comprovados em um corpo considerável de literatura científica. Agora, um novo estudo mostra que muito de uma coisa b
 Os inibidores GSK-3 mostram-se promissores no tratamento de infecções por coronavírus
Pesquisadores nos Estados Unidos sugeriram uma nova abordagem para tratar a infecção com coronavírus, como a síndrome respiratória aguda grave coronavírus 2 (SARS-CoV-2) - o agente que causa a doença
Os inibidores GSK-3 mostram-se promissores no tratamento de infecções por coronavírus
Pesquisadores nos Estados Unidos sugeriram uma nova abordagem para tratar a infecção com coronavírus, como a síndrome respiratória aguda grave coronavírus 2 (SARS-CoV-2) - o agente que causa a doença
 O tipo de bactéria das vias aéreas superiores pode influenciar a gravidade da asma
Um novo estudo levanta a possibilidade de modular os sintomas da asma por meio de bactérias que comumente vivem nas vias aéreas superiores. O trabalho, que foi publicado em 16 de dezembro, 2019, no jo
O tipo de bactéria das vias aéreas superiores pode influenciar a gravidade da asma
Um novo estudo levanta a possibilidade de modular os sintomas da asma por meio de bactérias que comumente vivem nas vias aéreas superiores. O trabalho, que foi publicado em 16 de dezembro, 2019, no jo