Para o estudo, publicado em 26 de maio em Célula , investigadores internacionais coletaram quase 5, 000 amostras ao longo de um período de três anos em 60 cidades em 32 países e seis continentes. Os pesquisadores analisaram as amostras usando uma técnica de sequenciamento genômico chamada sequenciamento shotgun para detectar a presença de vários micróbios, incluindo bactérias, archaea (organismos unicelulares distintos das bactérias), e vírus que usam DNA como seu material genético. (Outros tipos de vírus que usam RNA como seu material genético, como SARS-CoV-2, o vírus que causa COVID-19, não teria sido detectado com os métodos de análise de DNA usados neste estudo pré-pandêmico.)
Este campo de pesquisa tem implicações importantes para detectar surtos de infecções conhecidas e desconhecidas e para estudar a prevalência de micróbios resistentes a antibióticos em diferentes ambientes urbanos.
"Cada vez que você se senta no metrô, você provavelmente está viajando diariamente com uma espécie inteiramente nova, "disse o autor sênior, Dr. Christopher Mason, co-diretor da WorldQuant Initiative for Quantitative Prediction e professor de fisiologia e biofísica na Weill Cornell Medicine. O Dr. Mason também é cofundador e consultor pago da Biotia e Onegevity Health, e um palestrante pago para a WorldQuant LLC.
O estudo atual levou à descoberta de 10, 928 vírus e 748 bactérias que não estão presentes em nenhuma base de dados de referência.
O Dr. Mason fundou a MetaSUB (abreviação de Metagenômica e Metadesign de Metrôs e Biomas Urbanos) em 2015, junto com o Dr. Evan Afshin, que era então um estudante de graduação no Macaulay Honors College no Queens College e agora é um bolsista clínico em fisiologia e biofísica na Weill Cornell Medicine e um consultor pago pela Onegevity Health.
O estudo recém-lançado foi liderado pelos drs. Pedreiro, David Danko, um aluno de doutorado da Weill Cornell Graduate School no laboratório do Dr. Mason durante o estudo, e Daniela Bezdan, que era pesquisador associado em biomedicina computacional na Weill Cornell Medicine na época.
Ao coletar amostras de micróbios e analisar seus genes - conhecidos coletivamente como microbioma - os pesquisadores esperam aprender mais sobre a bactéria, vírus e outros microorganismos que vivem entre os humanos. Por exemplo, a pesquisa pode ajudar a identificar o surgimento de cepas resistentes a antibióticos.
Prever a resistência a antibióticos apenas a partir de sequências genéticas é um desafio, mas os pesquisadores foram capazes de mapear alguns genes conhecidos por estarem ligados à resistência, quantificar sua abundância e confirmar a capacidade dos marcadores genéticos de conferir resistência. Eles descobriram que algumas cidades tinham mais genes de resistência do que outras, e que pode haver assinaturas específicas da cidade para alguns desses genes.
A resistência antimicrobiana continua sendo um grande desafio para a saúde global. "Embora mais pesquisas sejam necessárias, este conjunto de dados demonstra o valor e o potencial do mapeamento e monitoramento do microbioma, e os insights que pode fornecer aos médicos, cientistas e funcionários da saúde pública, "Dr. Afshin disse.
Além disso, aprender sobre as pequenas moléculas e proteínas feitas por micróbios também pode levar à descoberta de novos antibióticos, bem como de outras moléculas com potencial para serem desenvolvidas como drogas. Muitos antibióticos e medicamentos atualmente em uso são derivados de fontes microbianas.
As descobertas feitas sobre novas espécies microbianas também podem levar a novas ferramentas e abordagens de laboratório, como novas maneiras de usar a ferramenta de edição molecular conhecida como CRISPR. Neste estudo, os pesquisadores descobriram 838, 532 novos arrays CRISPR - fragmentos de DNA viral encontrados dentro das bactérias - e 4,3 milhões de novos peptídeos (pequenas proteínas).
Devido a esses esforços de amostragem, O Dr. Mason disse que pode prever com cerca de 90 por cento de precisão onde uma pessoa mora, apenas sequenciando o DNA em seus sapatos. Muitos fatores foram encontrados para influenciar o microbioma de uma cidade, incluindo população geral e densidade populacional, elevação, proximidade do oceano e do clima. As descobertas sobre essas assinaturas distintas podem permitir estudos forenses futuros.
Um microbioma contém ecos moleculares do local onde foi coletado. Uma amostra costeira pode conter micróbios amantes do sal, enquanto uma amostra de uma cidade densamente povoada pode mostrar uma biodiversidade impressionante, "Dr. Danko disse.
Dr. David Danko, Estudante de Soctoral, Escola de Pós-Graduação Weill Cornell.
Drs. Mason e Afshin começaram a coletar e analisar amostras microbianas no sistema de metrô da cidade de Nova York em 2013. Depois de publicar suas primeiras descobertas, apelidado de PathoMap, eles foram contatados por pesquisadores de todo o mundo que queriam fazer estudos semelhantes para suas próprias cidades.
O interesse internacional inspirou o laboratório do Dr. Mason a criar o MetaSUB e recrutou Daniela Bezdan como diretora de pesquisa. "Precisávamos de protocolos aceitos internacionalmente, acordos de logística e colaboração com cientistas, vendedores, escritórios governamentais e fundações filantrópicas para potencialmente 100 cidades em 20 países, "Bezdan disse.
Hoje, a MetaSUB continua a crescer e se expandiu para coletar amostras de RNA e DNA do ar, água e esgoto, além de superfícies duras. Isso resultou em uma doação de US $ 5 milhões em sequenciamento de águas residuais e rastreamento viral em três estados (Flórida, Nova York e Wisconsin), e que faz parte do novo Sistema Nacional de Vigilância de Águas Residuais (NWSS) do Center for Disease Control and Prevention.
O grupo também supervisiona projetos como o Global City Sampling Day (gCSD), realizada todos os anos em 21 de junho, e fez estudos abrangentes, incluindo uma análise microbiana abrangente do Rio de Janeiro antes, durante e após os Jogos Olímpicos de 2016. Muitas das amostras analisadas no estudo atual foram coletadas no Global City Sampling Day em 2016 e 2017.
O esforço de amostragem da cidade de Nova York foi realizado com o apoio do Weill Cornell Medicine Clinical and Translational Science Center (CTSC), em colaboração com o gerente sênior do programa CTSC, Jeff Zhu. O Dr. Mason e seus colegas estão atualmente se preparando para o evento deste ano.
“Quando começamos em 2015, o consórcio era formado por 16 cidades; seis anos depois, temos mais de 100 cidades. É ótimo ter esse grupo de curiosos, co-investigadores entusiastas e iniciantes, "disse o Dr. Mason, que também é professor de genômica computacional em biomedicina computacional no Instituto de Biomedicina Computacional em Weill Cornell Medicine, Príncipe Alwaleed Bin Talal Bin Abdulaziz Al-Saud, HRH.
"Embora as amostras sejam coletadas em todo o mundo, grande parte da análise é feita aqui mesmo na cidade de Nova York, na Weill Cornell Medicine, "disse o Dr. Mason. A análise e montagem de sequências também alavancou Bridges e Bridges-2, Supercomputadores do Extreme Science and Engineering Discovery Environment (XSEDE) no Pittsburgh Supercomputing Center.
Pesquisadores da MetaSUB na Suíça (Drs. Andre Kahles e Gunnar Rätsch) usaram essas montagens e dados brutos para construir um pesquisável, portal de sequência global de DNA (MetaGraph) que indexou todas as sequências genéticas conhecidas (incluindo dados MetaSUB). O portal mapeia qualquer elemento genético conhecido ou recém-descoberto para sua localização na Terra e pode ajudar na descoberta de novas interações microbianas e funções putativas.
O isolamento de DNA de amostras foi realizado em grande parte com o apoio da Zymo Research e Promega, e sequenciado em colaboração com o Dr. Shawn Levy no HudsonAlpha Institute for Biotechnology, Dr. Klas Udekwu, da Universidade de Estocolmo e do New York Genome Center. Estudos futuros e em andamento examinarão o RNA e o DNA com leituras longas e métodos de imagem espacial, bem como rastrear os metabólitos dos sites globais, e continuar a atualizar o mapa genético em escala planetária.
 Os inibidores GSK-3 mostram-se promissores no tratamento de infecções por coronavírus
Os inibidores GSK-3 mostram-se promissores no tratamento de infecções por coronavírus
 Microbioma pulmonar prevê a gravidade da doença COVID-19
Microbioma pulmonar prevê a gravidade da doença COVID-19
 Dieta mediterrânea promove envelhecimento saudável com microbioma intestinal mais saudável
Dieta mediterrânea promove envelhecimento saudável com microbioma intestinal mais saudável
 Como fatores do hospedeiro, como o microbioma pulmonar, auxiliam na infecção por SARS ‐ CoV ‐ 2?
Como fatores do hospedeiro, como o microbioma pulmonar, auxiliam na infecção por SARS ‐ CoV ‐ 2?
 Uma nova pesquisa identifica uma ligação entre o microbioma intestinal e derrames
Uma nova pesquisa identifica uma ligação entre o microbioma intestinal e derrames
 IBD muito mais comum do que o esperado,
IBD muito mais comum do que o esperado,
 O pH ácido aumenta a infecção por SARS-CoV-2 pela regulação positiva do receptor ACE2
A pandemia de doença coronavírus em curso 2019 (COVID-19) que é causada por um novo coronavírus, nomeadamente, síndrome respiratória aguda grave coronavírus 2 (SARS-CoV-2), já ceifou mais de 4,6 milhõ
O pH ácido aumenta a infecção por SARS-CoV-2 pela regulação positiva do receptor ACE2
A pandemia de doença coronavírus em curso 2019 (COVID-19) que é causada por um novo coronavírus, nomeadamente, síndrome respiratória aguda grave coronavírus 2 (SARS-CoV-2), já ceifou mais de 4,6 milhõ
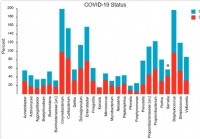 A composição e a estrutura do microbioma nasofaríngeo estão relacionadas à gravidade da doença COVID-19
As infecções virais estão associadas a alterações no microbioma do trato respiratório superior / nasofaríngeo (PN). Além disso, muitos estudos levantam a hipótese das possibilidades de superinfecções
A composição e a estrutura do microbioma nasofaríngeo estão relacionadas à gravidade da doença COVID-19
As infecções virais estão associadas a alterações no microbioma do trato respiratório superior / nasofaríngeo (PN). Além disso, muitos estudos levantam a hipótese das possibilidades de superinfecções
 Humanos versus vírus - podemos evitar a extinção em um futuro próximo?
O especialista argumenta que as mudanças no meio ambiente causadas pelo homem podem levar ao surgimento de patógenos, não apenas de fora, mas também de nosso próprio microbioma, que pode preparar o ca
Humanos versus vírus - podemos evitar a extinção em um futuro próximo?
O especialista argumenta que as mudanças no meio ambiente causadas pelo homem podem levar ao surgimento de patógenos, não apenas de fora, mas também de nosso próprio microbioma, que pode preparar o ca