A nova técnica de escrita de DNA, que os pesquisadores chamam de HiSCRIBE, é muito mais eficiente do que os sistemas desenvolvidos anteriormente para edição de DNA em bactérias, que teve uma taxa de sucesso de apenas cerca de 1 em 10, 000 células por geração. Em um novo estudo, os pesquisadores demonstraram que essa abordagem poderia ser usada para armazenar memória de interações celulares ou localização espacial.
Essa técnica também pode possibilitar a edição seletiva, ativar, ou silenciar genes em certas espécies de bactérias que vivem em uma comunidade natural, como o microbioma humano, dizem os pesquisadores.
Com este novo sistema de escrita de DNA, podemos editar genomas bacterianos com precisão e eficiência, sem a necessidade de qualquer forma de seleção, dentro de ecossistemas bacterianos complexos. Isso nos permite realizar a edição do genoma e a escrita do DNA fora dos ambientes de laboratório, seja para engenharia de bactérias, otimizar características de interesse in situ, ou estudar a dinâmica evolutiva e as interações nas populações bacterianas. "
Fahim Farzadfard, ex-pós-doutorado do MIT e principal autor do artigo
Timothy Lu, um professor associado do MIT de engenharia elétrica e ciência da computação e de engenharia biológica, é o autor sênior do estudo, que aparece hoje em Sistemas Celulares . Nava Gharaei, um ex-aluno de pós-graduação da Universidade de Harvard, e Robert Citorik, um ex-aluno de pós-graduação do MIT, também são autores do estudo.
Por muitos anos, O laboratório de Lu vem trabalhando em maneiras de usar o DNA para armazenar informações como a memória de eventos celulares. Em 2014, ele e Farzadfard desenvolveram uma maneira de empregar bactérias como um "gravador genômico, " Engenharia E. coli para armazenar memórias de eventos de longo prazo, como uma exposição a produtos químicos.
Para conseguir isso, os pesquisadores projetaram as células para produzir uma enzima transcriptase reversa chamada retron, que produz um DNA de fita simples (ssDNA) quando expresso nas células, e uma enzima recombinase, que pode inserir ("escrever") uma sequência específica de DNA de fita simples em um local-alvo no genoma. Este DNA é produzido apenas quando ativado pela presença de uma molécula pré-determinada ou outro tipo de entrada, como luz. Depois que o DNA é produzido, a recombinase insere o DNA em um local pré-programado, que pode estar em qualquer parte do genoma.
Essa técnica, que os pesquisadores chamaram de SCRIBE, teve uma eficiência de escrita relativamente baixa. Em cada geração, de 10, 000 E. coli células, apenas um adquiriria o novo DNA que os pesquisadores tentaram incorporar às células. Isso ocorre em parte porque o E. coli têm mecanismos celulares que evitam que o DNA de fita simples seja acumulado e integrado em seus genomas.
No novo estudo, os pesquisadores tentaram aumentar a eficiência do processo, eliminando alguns dos E. coli ' s mecanismos de defesa contra DNA de fita simples. Primeiro, eles desativaram enzimas chamadas exonucleases, que quebram o DNA de fita simples. Eles também eliminaram genes envolvidos em um sistema chamado de reparo de incompatibilidade, o que normalmente impede a integração do DNA de fita simples no genoma.
Com essas modificações, os pesquisadores conseguiram uma incorporação quase universal das mudanças genéticas que tentaram introduzir, criando uma maneira incomparável e eficiente para editar genomas bacterianos sem a necessidade de seleção.
"Por causa dessa melhora, fomos capazes de fazer algumas aplicações que não éramos capazes de fazer com a geração anterior do SCRIBE ou com outras tecnologias de escrita de DNA, "Farzadfard diz.
Em seu estudo de 2014, os pesquisadores mostraram que poderiam usar o SCRIBE para registrar a duração e a intensidade da exposição a uma molécula específica. Com seu novo sistema HiSCRIBE, eles podem rastrear esses tipos de exposições, bem como tipos adicionais de eventos, como interações entre células.
Por exemplo, os pesquisadores mostraram que podiam rastrear um processo chamado conjugação bacteriana, durante o qual as bactérias trocam pedaços de DNA. Ao integrar um "código de barras" de DNA no genoma de cada célula, que pode então ser trocada por outras células, os pesquisadores podem determinar quais células interagiram umas com as outras sequenciando seu DNA para ver quais códigos de barras elas carregam.
Esse tipo de mapeamento pode ajudar os pesquisadores a estudar como as bactérias se comunicam entre si em agregados, como biofilmes. Se uma abordagem semelhante pudesse ser implantada em células de mamíferos, poderia algum dia ser usado para mapear as interações entre outros tipos de células, como neurônios, Farzadfard diz. Os vírus que podem cruzar as sinapses neurais podem ser programados para transportar códigos de barras de DNA que os pesquisadores podem usar para rastrear conexões entre os neurônios, oferecendo uma nova maneira de ajudar a mapear o conectoma do cérebro.
“Estamos usando o DNA como mecanismo para registrar informações espaciais sobre a interação das células bacterianas, e talvez no futuro, neurônios que foram marcados, "Farzadfard diz.
Os pesquisadores também mostraram que poderiam usar essa técnica para editar especificamente o genoma de uma espécie de bactéria dentro de uma comunidade de muitas espécies. Nesse caso, eles introduziram o gene para uma enzima que quebra a galactose em E. coli células que crescem em cultura com várias outras espécies de bactérias.
Este tipo de edição seletiva de espécies pode oferecer uma nova maneira de tornar as bactérias resistentes a antibióticos mais suscetíveis aos medicamentos existentes, silenciando seus genes de resistência, dizem os pesquisadores. Contudo, tais tratamentos provavelmente exigiriam vários anos a mais anos de pesquisa para serem desenvolvidos, eles dizem.
Os pesquisadores também mostraram que poderiam usar essa técnica para criar um ecossistema sintético feito de bactérias e bacteriófagos que podem reescrever continuamente certos segmentos de seu genoma e evoluir de forma autônoma a uma taxa maior do que seria possível pela evolução natural. Nesse caso, eles foram capazes de otimizar a capacidade das células de consumir o consumo de lactose.
"Esta abordagem pode ser usada para a engenharia evolutiva de características celulares, ou em estudos de evolução experimental, permitindo que você repita a fita da evolução indefinidamente, "Farzadfard diz.
 Destaques e principais conclusões do Boston Bacterial Meeting (BBM) 2019
Destaques e principais conclusões do Boston Bacterial Meeting (BBM) 2019
 Se você tiver mais de 50 anos,
Se você tiver mais de 50 anos,
 Um novo receptor de macrófago superativador poderia explicar a hiperinflamação em COVID-19 grave
Um novo receptor de macrófago superativador poderia explicar a hiperinflamação em COVID-19 grave
 A pesquisa mostra como os micróbios intestinais afetam a gripe estomacal
A pesquisa mostra como os micróbios intestinais afetam a gripe estomacal
 Cientistas desenvolvem pílula impressa em 3D que coleta amostras de bactérias encontradas no intestino
Cientistas desenvolvem pílula impressa em 3D que coleta amostras de bactérias encontradas no intestino
 Inflamação intestinal limitada em COVID-19
Inflamação intestinal limitada em COVID-19
 Vazamento intestinal e disbiose microbiana podem contribuir para a tempestade de citocinas em casos de COVID-19 gravemente enfermos
À medida que o mundo se aproxima do marco sombrio de três milhões de mortes pela doença COVID-19, um novo artigo de pesquisa de pré-impressão publicado no bioRxiv * servidor mostra que a presença de
Vazamento intestinal e disbiose microbiana podem contribuir para a tempestade de citocinas em casos de COVID-19 gravemente enfermos
À medida que o mundo se aproxima do marco sombrio de três milhões de mortes pela doença COVID-19, um novo artigo de pesquisa de pré-impressão publicado no bioRxiv * servidor mostra que a presença de
 Destaques e principais conclusões do Boston Bacterial Meeting (BBM) 2019
O Boston Bacterial Meeting (BBM) é uma conferência anual que reúne especialistas em bacteriologia da área de Boston e de todo o mundo. São 25 º iteração ocorreu entre 6 de junho º e 7 de junho º
Destaques e principais conclusões do Boston Bacterial Meeting (BBM) 2019
O Boston Bacterial Meeting (BBM) é uma conferência anual que reúne especialistas em bacteriologia da área de Boston e de todo o mundo. São 25 º iteração ocorreu entre 6 de junho º e 7 de junho º
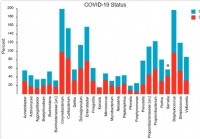 A composição e a estrutura do microbioma nasofaríngeo estão relacionadas à gravidade da doença COVID-19
As infecções virais estão associadas a alterações no microbioma do trato respiratório superior / nasofaríngeo (PN). Além disso, muitos estudos levantam a hipótese das possibilidades de superinfecções
A composição e a estrutura do microbioma nasofaríngeo estão relacionadas à gravidade da doença COVID-19
As infecções virais estão associadas a alterações no microbioma do trato respiratório superior / nasofaríngeo (PN). Além disso, muitos estudos levantam a hipótese das possibilidades de superinfecções