O dispositivo também pode ser usado para detectar mutações virais específicas ligadas a algumas das variantes do SARS-CoV-2 que agora estão circulando. Este resultado também pode ser obtido dentro de uma hora, potencialmente tornando muito mais fácil rastrear diferentes variantes do vírus, especialmente em regiões que não têm acesso a instalações de sequenciamento genético.
Demonstramos que nossa plataforma pode ser programada para detectar novas variantes que surgem, e que poderíamos redirecioná-lo rapidamente. Neste estudo, visamos o Reino Unido, África do Sul, e variantes brasileiras, mas você pode adaptar prontamente a plataforma de diagnóstico para lidar com a variante Delta e outras que estão surgindo. "
James Collins, o professor Termeer de Engenharia Médica e Ciência no Instituto de Engenharia Médica e Ciência do MIT (IMES) e no Departamento de Engenharia Biológica
O novo diagnóstico, que depende da tecnologia CRISPR, pode ser montado por cerca de US $ 15, mas esses custos podem diminuir significativamente se os dispositivos forem produzidos em grande escala, dizem os pesquisadores.
Collins é o autor sênior do novo estudo, que aparece hoje em Avanços da Ciência . Os principais autores do artigo são Helena de Puig, pós-doutorado no Instituto Wyss de Engenharia Inspirada na Biologia da Universidade de Harvard; Rose Lee, instrutor de pediatria no Hospital Infantil de Boston e no Centro Médico Beth Israel Deaconess e pesquisador visitante no Instituto Wyss; Devora Najjar, um estudante de pós-graduação no Media Lab do MIT; e Xiao Tan, bolsista clínico do Wyss Institute e instrutor de gastroenterologia no Massachusetts General Hospital.
O novo diagnóstico é baseado em SHERLOCK, uma ferramenta baseada em CRISPR que Collins e outros relataram pela primeira vez em 2017. Os componentes do sistema incluem uma fita guia de RNA que permite a detecção de sequências específicas de RNA alvo, e enzimas Cas que clivam essas sequências e produzem um sinal fluorescente. Todos esses componentes moleculares podem ser liofilizados para armazenamento de longo prazo e reativados mediante exposição à água.
Ano passado, O laboratório de Collins começou a trabalhar na adaptação desta tecnologia para detectar o vírus SARS-CoV-2, esperando que eles pudessem projetar um dispositivo de diagnóstico que pudesse produzir resultados rápidos e ser operado com pouca ou nenhuma experiência. Eles também queriam que funcionasse com amostras de saliva, tornando ainda mais fácil para os usuários.
Para conseguir isso, os pesquisadores tiveram que incorporar uma etapa crítica de pré-processamento que desabilita enzimas chamadas nucleases salivares, que destroem ácidos nucléicos como o RNA. Uma vez que a amostra vai para o dispositivo, as nucleases são inativadas pelo calor e dois reagentes químicos. Então, O RNA viral é extraído e concentrado ao passar a saliva através de uma membrana.
"Essa membrana foi a chave para coletar os ácidos nucléicos e concentrá-los para que possamos obter a sensibilidade que estamos mostrando com este diagnóstico, "Lee diz.
Esta amostra de RNA é então exposta a componentes CRISPR / Cas liofilizados, que são ativados por perfuração automática de pacotes de água selados dentro do dispositivo. A reação one-pot amplifica a amostra de RNA e, em seguida, detecta a sequência de RNA alvo, se presente.
"Nosso objetivo era criar um diagnóstico totalmente autocontido que não requer outro equipamento, "Tan diz." Essencialmente, o paciente cospe neste dispositivo, e então você pressiona um êmbolo e obtém uma resposta uma hora depois. "
Os pesquisadores projetaram o dispositivo, que eles chamam de SHERLOCK minimamente instrumentado (miSHERLOCK), de modo que pode ter até quatro módulos, cada um procurando uma sequência diferente de RNA alvo. O módulo original contém fitas-guia de RNA que detectam qualquer cepa de SARS-CoV-2. Outros módulos são específicos para mutações associadas a algumas das variantes que surgiram no ano passado, incluindo B.1.1.7, P.1, e B.1.351.
A variante Delta ainda não estava difundida quando os pesquisadores realizaram este estudo, mas porque o sistema já está construído, eles dizem que deve ser simples projetar um novo módulo para detectar essa variante. O sistema também pode ser facilmente programado para monitorar novas mutações que podem tornar o vírus mais infeccioso.
“Se você quiser fazer um levantamento epidemiológico mais amplo, você pode projetar ensaios antes que uma mutação de interesse apareça em uma população, para monitorar mutações potencialmente perigosas na proteína do pico, "Najjar diz.
Os pesquisadores testaram seu dispositivo pela primeira vez com saliva humana enriquecida com sequências de RNA sintético SARS-CoV-2, e depois com cerca de 50 amostras de pacientes com teste positivo para o vírus. Eles descobriram que o dispositivo era tão preciso quanto os testes de PCR padrão ouro usados agora, que requerem cotonetes nasais e levam mais tempo e significativamente mais hardware e manipulação de amostras para produzir resultados.
O dispositivo produz uma leitura fluorescente que pode ser vista a olho nu, e os pesquisadores também desenvolveram um aplicativo para smartphone que pode ler os resultados e enviá-los aos departamentos de saúde pública para facilitar o rastreamento.
Os pesquisadores acreditam que seu dispositivo pode ser produzido a um custo tão baixo quanto $ 2 a $ 3 por dispositivo. Se aprovado pelo FDA e fabricado em grande escala, eles imaginam que este tipo de diagnóstico pode ser útil para pessoas que querem ser capazes de fazer o teste em casa, ou em centros de saúde em áreas sem amplo acesso a testes de PCR ou sequenciamento genético de variantes do SARS-CoV-2.
"A capacidade de detectar e rastrear essas variantes é essencial para uma saúde pública eficaz, mas infelizmente, variantes são atualmente diagnosticadas apenas por sequenciamento de ácido nucleico em centros epidemiológicos especializados que são escassos, mesmo em nações ricas em recursos, "de Puig diz.
 Cientistas desenvolvem peptídeos que restauram o equilíbrio das bactérias intestinais e revertem a aterosclerose
Cientistas desenvolvem peptídeos que restauram o equilíbrio das bactérias intestinais e revertem a aterosclerose
 Os probióticos podem ajudar a conter a desnutrição nas próximas duas décadas,
Os probióticos podem ajudar a conter a desnutrição nas próximas duas décadas,
 A superbactéria E. coli se espalhando por falta de higiene do banheiro,
A superbactéria E. coli se espalhando por falta de higiene do banheiro,
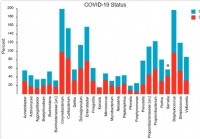 A composição e a estrutura do microbioma nasofaríngeo estão relacionadas à gravidade da doença COVID-19
A composição e a estrutura do microbioma nasofaríngeo estão relacionadas à gravidade da doença COVID-19
 A disbiose na microbiota intestinal pode causar infecção secundária grave em pacientes com COVID-19
A disbiose na microbiota intestinal pode causar infecção secundária grave em pacientes com COVID-19
 Sangue jovem restaura vitalidade em idosos
Sangue jovem restaura vitalidade em idosos
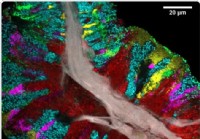 Mapa detalhado do microbioma da língua humana
Um novo estudo publicado na revista Relatórios de Célula em março de 2020, relata o uso de técnicas de imagem espectrais avançadas e rápidas para desenvolver um mapa detalhado das comunidades microb
Mapa detalhado do microbioma da língua humana
Um novo estudo publicado na revista Relatórios de Célula em março de 2020, relata o uso de técnicas de imagem espectrais avançadas e rápidas para desenvolver um mapa detalhado das comunidades microb
 O café ajuda a desenvolver micróbios intestinais saudáveis e evita os movimentos intestinais
Houve evidências anedóticas de que os bebedores de café juram que sua xícara matinal apresenta movimentos intestinais normais pela manhã. Um novo estudo de pesquisadores no Texas mostrou que o café re
O café ajuda a desenvolver micróbios intestinais saudáveis e evita os movimentos intestinais
Houve evidências anedóticas de que os bebedores de café juram que sua xícara matinal apresenta movimentos intestinais normais pela manhã. Um novo estudo de pesquisadores no Texas mostrou que o café re
 A vibração de todo o corpo ajuda a reduzir a inflamação,
graças ao microbioma intestinal A vibração de todo o corpo parece melhorar muitos sintomas da diabetes mellitus tipo II, em que a glicose e a inflamação destrutiva disparam. O procedimento ajuda o cor
A vibração de todo o corpo ajuda a reduzir a inflamação,
graças ao microbioma intestinal A vibração de todo o corpo parece melhorar muitos sintomas da diabetes mellitus tipo II, em que a glicose e a inflamação destrutiva disparam. O procedimento ajuda o cor